Aritalab:Lecture/Biochem/Protein
m |
|||
| Line 1: | Line 1: | ||
| − | |||
| − | |||
{| style="float:right" | {| style="float:right" | ||
| __TOC__ | | __TOC__ | ||
Revision as of 11:54, 24 April 2012
|
タンパク質は20種類のアミノ酸が連なったポリペプチドです。
タンパク質を大きく分けると表のようになります。
| 水溶性タンパク | 球状 (globular) | 酵素や転写因子など。分子内部は疎水性、外部は親水性。 |
| 不定形 (disordered) | 真核生物に多い不定構造。親水性アミノ酸が多い。とりわけEPQSRKを含む。 | |
| 膜タンパク | 受容体、トランスポーターなど。疎水性アミノ酸が多い。とりわけ膜貫通領域は疎水性。結晶化しにくい。 | |
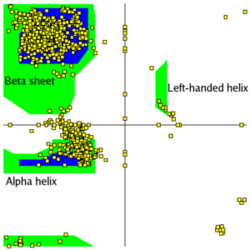
ラマチャンドランプロット
コンタクトマップ
縦(上から下にN→C)横(左から右にN→C)にペプチド鎖を並べ、Cα原子間の距離が10オングストローム以内であれば色を塗ったものをコンタクトマップと呼びます。 対角線は同一の残基がくるので黒くなります。α-へリックスはこの対角線上に重なる形で表示されます。 平行β-シートは対角線から離れた位置に同じ角度で現れ、逆平行β-シートは、対角線と直行する角度で現れます。
構造の分類と測定
タンパク質立体構造の研究はミオグロビン(myoglobin: 筋肉にある赤いタンパク質)から始まりました。ジョン・ケンドリュー(John Kendrew)がX線回折で世界ではじめてタンパク質の構造を明らかにしたのは1960年です(そんなに昔ではないのです)。立体構造解析に対する功績により、ケンドリューはわずか2年後の1962年にノーベル化学賞を授与されています(ペルーツと共同)。
DNAに比較すると、タンパク質の構造は遥かに複雑で理解が難しいものでした。
ドメインによる分類
タンパク質の構造は、長さが 50-150アミノ酸程度のドメインと呼ばれるブロックに分けて考えます。各ドメインは機能部位や疎水性コアを持ち、ドメイン間の二次構造どうしは近接しません。ドメインとはつまり、タンパク質の構造(および機能)モジュールと捉えられます。金属イオンと結合して構造を形成するドメイン(例. ジンクフィンガー)もあります。
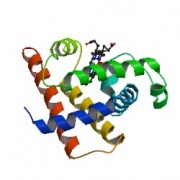
立体構造を分類するデータベースには、手作業で構造を分けた SCOP (structural classification of proteins) や CATH があります。いずれもドメインを考慮した分類を採用しており、大きく分けると以下のようになります。(図はCATH DBより)
mainly α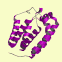
|
mainly β
|
α and β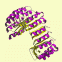
|
few structures
|
測定方法
- ゲル電気泳動
タンパク質は帯電しているので、ポリアクリルアミド電気泳動 (PAGE) で分離できます。
- X線回折
タンパク質を結晶化させられる場合はX線結晶解析により原子位置を特定できます。 粒子加速器から得られるX線を用いると、Cαほか、重い原子の位置を計算できます。水素の位置はわかりません。
- NMR解析
NMRとはNuclear Magnetic Resonanceの略で、原子核の磁気共鳴を用いて原子の位置を特定します。
- 解説、参考