Mol:FLIA2CGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 50 0 0 0 0 0 0 0 0999 V2000 | + | 45 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3915 0.9213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3915 0.9213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1648 0.6001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1648 0.6001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7211 0.9213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7211 0.9213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2772 0.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2772 0.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2772 -0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2772 -0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8539 -0.3987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8539 -0.3987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4307 -0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4307 -0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4307 0.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4307 0.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8539 0.9332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8539 0.9332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1650 -0.0419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1650 -0.0419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7211 -0.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7211 -0.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8539 -1.0642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8539 -1.0642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0070 -0.3985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0070 -0.3985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0070 -1.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0070 -1.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5584 -1.3534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5584 -1.3534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1097 -1.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1097 -1.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1097 -0.3985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1097 -0.3985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5584 -0.0801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5584 -0.0801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4954 0.7175 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.4954 0.7175 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.1492 0.2605 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1492 0.2605 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6506 0.4544 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6506 0.4544 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1309 0.4486 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1309 0.4486 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5191 0.8092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5191 0.8092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0284 0.6264 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0284 0.6264 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.8824 0.9410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8824 0.9410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4760 -0.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4760 -0.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3649 -0.0252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3649 -0.0252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5084 -1.9317 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5084 -1.9317 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.9357 -1.9602 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9357 -1.9602 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7638 -1.4536 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7638 -1.4536 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.4253 -1.0593 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4253 -1.0593 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9510 -1.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9510 -1.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1384 -1.6325 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.1384 -1.6325 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.8243 -2.2477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8243 -2.2477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8161 -2.4871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8161 -2.4871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2128 -1.5431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2128 -1.5431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7152 -1.2319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7152 -1.2319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0894 -0.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0894 -0.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7152 -0.2017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7152 -0.2017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2980 1.3534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2980 1.3534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6599 2.1233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6599 2.1233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8686 -1.3718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8686 -1.3718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6308 -2.0191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6308 -2.0191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5495 0.0501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5495 0.0501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5495 -0.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5495 -0.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 16 37 1 0 0 0 0 | + | 16 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 17 1 0 0 0 0 | + | 39 17 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 10 44 1 0 0 0 0 | + | 10 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 42 43 | + | M SAL 3 2 42 43 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 47 -4.5048 0.5284 | + | M SVB 3 47 -4.5048 0.5284 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 45 -2.298 1.3534 | + | M SVB 2 45 -2.298 1.3534 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 49 | + | M SBL 1 1 49 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 49 -0.5495 0.0501 | + | M SVB 1 49 -0.5495 0.0501 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIA2CGS0002 | + | ID FLIA2CGS0002 |
| − | KNApSAcK_ID C00010098 | + | KNApSAcK_ID C00010098 |
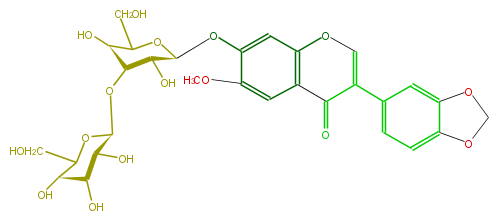
| − | NAME Fujikinetin 7-O-laminaribioside | + | NAME Fujikinetin 7-O-laminaribioside |
| − | CAS_RN 68862-14-6 | + | CAS_RN 68862-14-6 |
| − | FORMULA C29H32O16 | + | FORMULA C29H32O16 |
| − | EXACTMASS 636.1690349759999 | + | EXACTMASS 636.1690349759999 |
| − | AVERAGEMASS 636.5547799999999 | + | AVERAGEMASS 636.5547799999999 |
| − | SMILES c(OC)(c(O[C@@H]([C@H]5O)OC(CO)[C@@H]([C@@H]5O[C@@H]([C@H]6O)OC([C@@H]([C@H](O)6)O)CO)O)4)cc(c(c4)1)C(=O)C(c(c3)cc(O2)c(c3)OC2)=CO1 | + | SMILES c(OC)(c(O[C@@H]([C@H]5O)OC(CO)[C@@H]([C@@H]5O[C@@H]([C@H]6O)OC([C@@H]([C@H](O)6)O)CO)O)4)cc(c(c4)1)C(=O)C(c(c3)cc(O2)c(c3)OC2)=CO1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 50 0 0 0 0 0 0 0 0999 V2000
-0.3915 0.9213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1648 0.6001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7211 0.9213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2772 0.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2772 -0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8539 -0.3987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4307 -0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4307 0.6002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8539 0.9332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1650 -0.0419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7211 -0.3630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8539 -1.0642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0070 -0.3985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0070 -1.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5584 -1.3534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1097 -1.0351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1097 -0.3985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5584 -0.0801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4954 0.7175 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.1492 0.2605 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6506 0.4544 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1309 0.4486 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5191 0.8092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0284 0.6264 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.8824 0.9410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4760 -0.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3649 -0.0252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5084 -1.9317 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.9357 -1.9602 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7638 -1.4536 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.4253 -1.0593 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9510 -1.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1384 -1.6325 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.8243 -2.2477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8161 -2.4871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2128 -1.5431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7152 -1.2319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0894 -0.7168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7152 -0.2017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2980 1.3534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6599 2.1233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8686 -1.3718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6308 -2.0191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5495 0.0501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5495 -0.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
28 29 1 1 0 0 0
29 30 1 1 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 26 1 0 0 0 0
16 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 17 1 0 0 0 0
24 40 1 0 0 0 0
40 41 1 0 0 0 0
33 42 1 0 0 0 0
42 43 1 0 0 0 0
10 44 1 0 0 0 0
44 45 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 42 43
M SBL 3 1 47
M SMT 3 CH2OH
M SVB 3 47 -4.5048 0.5284
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 CH2OH
M SVB 2 45 -2.298 1.3534
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 49
M SMT 1 OCH3
M SVB 1 49 -0.5495 0.0501
S SKP 8
ID FLIA2CGS0002
KNApSAcK_ID C00010098
NAME Fujikinetin 7-O-laminaribioside
CAS_RN 68862-14-6
FORMULA C29H32O16
EXACTMASS 636.1690349759999
AVERAGEMASS 636.5547799999999
SMILES c(OC)(c(O[C@@H]([C@H]5O)OC(CO)[C@@H]([C@@H]5O[C@@H]([C@H]6O)OC([C@@H]([C@H](O)6)O)CO)O)4)cc(c(c4)1)C(=O)C(c(c3)cc(O2)c(c3)OC2)=CO1
M END