Mol:FL7AAGGA0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5144 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5144 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5144 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5144 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7999 0.3152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7999 0.3152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0855 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0855 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0855 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0855 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7999 1.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7999 1.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6289 0.3152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6289 0.3152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3434 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3434 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3434 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3434 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6289 1.9651 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.6289 1.9651 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1199 2.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1199 2.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8343 1.5884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8343 1.5884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5488 2.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5488 2.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5488 2.8259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5488 2.8259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8343 3.2384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8343 3.2384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1199 2.8259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1199 2.8259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1890 3.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1890 3.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2019 0.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2019 0.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0944 1.8875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0944 1.8875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7999 -0.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7999 -0.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8343 3.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8343 3.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2138 1.6170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2138 1.6170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0609 -1.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0609 -1.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8664 -1.8831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8664 -1.8831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2519 -1.1534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2519 -1.1534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9554 -0.7220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9554 -0.7220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1498 -0.5429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1498 -0.5429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7642 -1.2726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7642 -1.2726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3529 -1.0570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3529 -1.0570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0637 -1.3488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0637 -1.3488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2086 -2.3700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2086 -2.3700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0420 -1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0420 -1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3510 -3.3627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3510 -3.3627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4341 -3.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4341 -3.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8868 -2.9272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8868 -2.9272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6280 -2.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6280 -2.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8430 -2.3098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8430 -2.3098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3901 -2.9997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3901 -2.9997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7720 -2.9826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7720 -2.9826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1281 -3.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1281 -3.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6968 -3.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6968 -3.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4715 -1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4715 -1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5376 0.0241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5376 0.0241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1250 -0.6905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1250 -0.6905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3267 -0.4813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3267 -0.4813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5332 -0.7082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5332 -0.7082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9457 0.0065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9457 0.0065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7441 -0.2025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7441 -0.2025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8730 -1.1700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8730 -1.1700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7844 -0.5881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7844 -0.5881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1074 -0.1809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1074 -0.1809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0030 0.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0030 0.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6378 0.4224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6378 0.4224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6364 1.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6364 1.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2138 1.4963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2138 1.4963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0752 1.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0752 1.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 26 18 1 0 0 0 0 | + | 26 18 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | 42 29 1 0 0 0 0 | + | 42 29 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 43 51 1 0 0 0 0 | + | 43 51 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 46 20 1 0 0 0 0 | + | 46 20 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAGGA0015 | + | ID FL7AAGGA0015 |
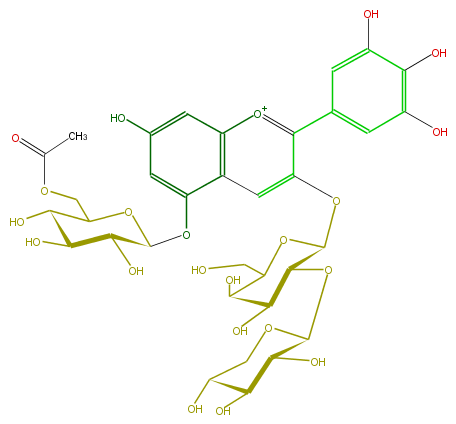
| − | FORMULA C34H41O22 | + | FORMULA C34H41O22 |
| − | EXACTMASS 801.208947996 | + | EXACTMASS 801.208947996 |
| − | AVERAGEMASS 801.67614 | + | AVERAGEMASS 801.67614 |
| − | SMILES OC(C6O)C(OC(COC(C)=O)C6O)Oc(c14)cc(O)cc1[o+1]c(c(c5)cc(c(c5O)O)O)c(c4)OC(C(OC(C3O)OCC(C3O)O)2)OC(C(C(O)2)O)CO | + | SMILES OC(C6O)C(OC(COC(C)=O)C6O)Oc(c14)cc(O)cc1[o+1]c(c(c5)cc(c(c5O)O)O)c(c4)OC(C(OC(C3O)OCC(C3O)O)2)OC(C(C(O)2)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-1.5144 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5144 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7999 0.3152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0855 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0855 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7999 1.9651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6289 0.3152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3434 0.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3434 1.5526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6289 1.9651 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.1199 2.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8343 1.5884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5488 2.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5488 2.8259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8343 3.2384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1199 2.8259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1890 3.1956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2019 0.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0944 1.8875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7999 -0.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8343 3.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2138 1.6170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0609 -1.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8664 -1.8831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2519 -1.1534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9554 -0.7220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1498 -0.5429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7642 -1.2726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3529 -1.0570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0637 -1.3488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2086 -2.3700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0420 -1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3510 -3.3627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4341 -3.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8868 -2.9272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6280 -2.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8430 -2.3098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3901 -2.9997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7720 -2.9826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1281 -3.9697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6968 -3.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4715 -1.1592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5376 0.0241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1250 -0.6905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3267 -0.4813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5332 -0.7082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9457 0.0065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7441 -0.2025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8730 -1.1700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7844 -0.5881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1074 -0.1809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0030 0.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6378 0.4224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6364 1.1317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2138 1.4963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0752 1.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
26 18 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
35 39 1 0 0 0 0
34 40 1 0 0 0 0
33 41 1 0 0 0 0
36 32 1 0 0 0 0
42 29 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
45 49 1 0 0 0 0
44 50 1 0 0 0 0
43 51 1 0 0 0 0
48 52 1 0 0 0 0
52 53 1 0 0 0 0
46 20 1 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
M CHG 1 10 1
S SKP 5
ID FL7AAGGA0015
FORMULA C34H41O22
EXACTMASS 801.208947996
AVERAGEMASS 801.67614
SMILES OC(C6O)C(OC(COC(C)=O)C6O)Oc(c14)cc(O)cc1[o+1]c(c(c5)cc(c(c5O)O)O)c(c4)OC(C(OC(C3O)OCC(C3O)O)2)OC(C(C(O)2)O)CO
M END