Mol:FL7AAAGL0056
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9854 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9854 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9854 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9854 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2709 -0.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2709 -0.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4435 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4435 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4435 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4435 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2709 1.4826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2709 1.4826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1580 -0.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1580 -0.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8725 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8725 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8725 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8725 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1580 1.4826 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 1.1580 1.4826 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6490 1.5184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6490 1.5184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3635 1.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3635 1.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0780 1.5184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0780 1.5184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0780 2.3434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0780 2.3434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3635 2.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3635 2.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6490 2.3434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6490 2.3434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7182 2.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7182 2.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5654 1.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5654 1.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2709 -0.9301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2709 -0.9301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5227 -0.1303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5227 -0.1303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2748 -2.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2748 -2.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0794 -2.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0794 -2.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4689 -1.5288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4689 -1.5288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1748 -1.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1748 -1.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3701 -0.9178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3701 -0.9178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9806 -1.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9806 -1.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4443 -1.3622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4443 -1.3622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9149 -1.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9149 -1.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3006 -2.7559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3006 -2.7559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5844 -2.4093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5844 -2.4093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8216 -2.0897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8216 -2.0897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2350 1.4121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2350 1.4121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8223 0.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8223 0.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0240 0.9066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0240 0.9066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2305 0.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2305 0.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6431 1.3945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6431 1.3945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4415 1.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4415 1.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5984 1.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5984 1.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0783 2.0483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0783 2.0483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7182 0.9289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7182 0.9289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2710 0.9565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2710 0.9565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1955 0.2666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1955 0.2666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 18 35 1 0 0 0 0 | + | 18 35 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0056 | + | ID FL7AAAGL0056 |
| − | KNApSAcK_ID C00014832 | + | KNApSAcK_ID C00014832 |
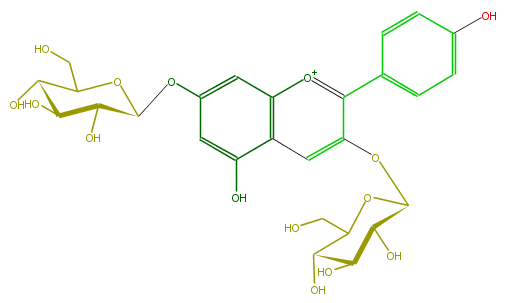
| − | NAME Pelargonidin 3,7-di-glucoside | + | NAME Pelargonidin 3,7-di-glucoside |
| − | CAS_RN 102521-86-8 | + | CAS_RN 102521-86-8 |
| − | FORMULA C27H31O15 | + | FORMULA C27H31O15 |
| − | EXACTMASS 595.166295322 | + | EXACTMASS 595.166295322 |
| − | AVERAGEMASS 595.52604 | + | AVERAGEMASS 595.52604 |
| − | SMILES C(C1O)(O)C(C(OC(Oc(c(c(c5)ccc(O)c5)2)cc(c(O)3)c(cc(OC(C(O)4)OC(CO)C(O)C4O)c3)[o+1]2)1)CO)O | + | SMILES C(C1O)(O)C(C(OC(Oc(c(c(c5)ccc(O)c5)2)cc(c(O)3)c(cc(OC(C(O)4)OC(CO)C(O)C4O)c3)[o+1]2)1)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.9854 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9854 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2709 -0.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4435 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4435 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2709 1.4826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1580 -0.1674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8725 0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8725 1.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1580 1.4826 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.6490 1.5184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3635 1.1059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0780 1.5184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0780 2.3434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3635 2.7559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6490 2.3434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7182 2.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5654 1.4050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2709 -0.9301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5227 -0.1303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2748 -2.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0794 -2.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4689 -1.5288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1748 -1.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3701 -0.9178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9806 -1.6454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4443 -1.3622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9149 -1.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3006 -2.7559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5844 -2.4093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8216 -2.0897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2350 1.4121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8223 0.6974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0240 0.9066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2305 0.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6431 1.3945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4415 1.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5984 1.7712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0783 2.0483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7182 0.9289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2710 0.9565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1955 0.2666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 20 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
18 35 1 0 0 0 0
S SKP 8
ID FL7AAAGL0056
KNApSAcK_ID C00014832
NAME Pelargonidin 3,7-di-glucoside
CAS_RN 102521-86-8
FORMULA C27H31O15
EXACTMASS 595.166295322
AVERAGEMASS 595.52604
SMILES C(C1O)(O)C(C(OC(Oc(c(c(c5)ccc(O)c5)2)cc(c(O)3)c(cc(OC(C(O)4)OC(CO)C(O)C4O)c3)[o+1]2)1)CO)O
M END