Mol:FL63ACGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.3791 -1.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3791 -1.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3791 -1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3791 -1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1389 -2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1389 -2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6569 -1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6569 -1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6569 -1.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6569 -1.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1389 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1389 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1749 -2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1749 -2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6929 -1.8158 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.6929 -1.8158 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6929 -1.2176 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.6929 -1.2176 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.1749 -0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1749 -0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2109 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2109 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7332 -1.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7332 -1.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2555 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2555 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2555 -0.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2555 -0.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7332 -0.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7332 -0.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2109 -0.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2109 -0.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7554 -0.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7554 -0.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8971 -0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8971 -0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2109 -2.1148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2109 -2.1148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7332 0.5883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7332 0.5883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0953 -1.1175 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.0953 -1.1175 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.7241 -1.6075 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7241 -1.6075 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1896 -1.3996 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1896 -1.3996 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6739 -1.3940 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6739 -1.3940 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.0486 -1.0192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0486 -1.0192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5162 -1.2660 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.5162 -1.2660 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.7554 -1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7554 -1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3027 -1.6356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3027 -1.6356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8833 -1.9137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8833 -1.9137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1389 -2.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1389 -2.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7692 2.2283 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.7692 2.2283 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.5304 1.6619 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5304 1.6619 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9777 1.3030 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9777 1.3030 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2404 0.8591 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.2404 0.8591 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3777 1.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3777 1.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9301 1.6527 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9301 1.6527 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.2860 2.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2860 2.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2167 2.1489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2167 2.1489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6856 0.7807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6856 0.7807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7224 -0.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7224 -0.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6768 -0.0476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6768 -0.0476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5471 2.5726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5471 2.5726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2616 2.1601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2616 2.1601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 17 14 1 0 0 0 0 | + | 17 14 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 20 34 1 0 0 0 0 | + | 20 34 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 2.5471 2.5726 | + | M SVB 2 46 2.5471 2.5726 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -2.7224 -0.3461 | + | M SVB 1 44 -2.7224 -0.3461 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACGS0023 | + | ID FL63ACGS0023 |
| − | KNApSAcK_ID C00008859 | + | KNApSAcK_ID C00008859 |
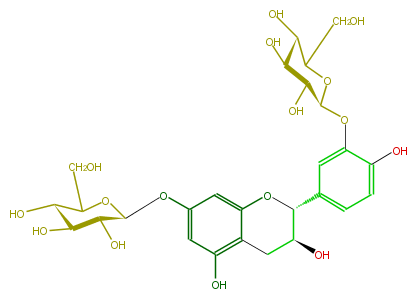
| − | NAME Catechin 7,3'-di-O-beta-D-glucopyranoside | + | NAME Catechin 7,3'-di-O-beta-D-glucopyranoside |
| − | CAS_RN 105330-53-8 | + | CAS_RN 105330-53-8 |
| − | FORMULA C27H34O16 | + | FORMULA C27H34O16 |
| − | EXACTMASS 614.18468504 | + | EXACTMASS 614.18468504 |
| − | AVERAGEMASS 614.54926 | + | AVERAGEMASS 614.54926 |
| − | SMILES [C@@H](O)([C@H]5O)[C@@H]([C@@H](OC(CO)5)Oc(c1)cc(O2)c(C[C@@H]([C@@H](c(c3)cc(O[C@H](O4)[C@H]([C@H]([C@@H](O)C(CO)4)O)O)c(O)c3)2)O)c1O)O | + | SMILES [C@@H](O)([C@H]5O)[C@@H]([C@@H](OC(CO)5)Oc(c1)cc(O2)c(C[C@@H]([C@@H](c(c3)cc(O[C@H](O4)[C@H]([C@H]([C@@H](O)C(CO)4)O)O)c(O)c3)2)O)c1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-0.3791 -1.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3791 -1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1389 -2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6569 -1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6569 -1.2176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1389 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1749 -2.1148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6929 -1.8158 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6929 -1.2176 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.1749 -0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2109 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7332 -1.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2555 -0.9186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2555 -0.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7332 -0.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2109 -0.3155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7554 -0.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8971 -0.9186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2109 -2.1148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7332 0.5883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0953 -1.1175 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.7241 -1.6075 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1896 -1.3996 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6739 -1.3940 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.0486 -1.0192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5162 -1.2660 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.7554 -1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3027 -1.6356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8833 -1.9137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1389 -2.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7692 2.2283 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.5304 1.6619 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9777 1.3030 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2404 0.8591 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3777 1.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9301 1.6527 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.2860 2.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2167 2.1489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6856 0.7807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7224 -0.3461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6768 -0.0476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5471 2.5726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2616 2.1601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
17 14 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
3 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
20 34 1 0 0 0 0
26 40 1 0 0 0 0
40 41 1 0 0 0 0
36 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 2.5471 2.5726
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -2.7224 -0.3461
S SKP 8
ID FL63ACGS0023
KNApSAcK_ID C00008859
NAME Catechin 7,3'-di-O-beta-D-glucopyranoside
CAS_RN 105330-53-8
FORMULA C27H34O16
EXACTMASS 614.18468504
AVERAGEMASS 614.54926
SMILES [C@@H](O)([C@H]5O)[C@@H]([C@@H](OC(CO)5)Oc(c1)cc(O2)c(C[C@@H]([C@@H](c(c3)cc(O[C@H](O4)[C@H]([C@H]([C@@H](O)C(CO)4)O)O)c(O)c3)2)O)c1O)O
M END