Mol:FL5FFLGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.7972 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7972 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0827 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0827 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0827 2.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0827 2.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7972 3.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7972 3.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5117 2.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5117 2.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5117 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5117 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3682 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3682 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6538 1.8364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6538 1.8364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0607 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0607 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0607 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0607 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6538 0.1864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6538 0.1864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3682 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3682 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7752 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7752 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4896 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4896 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4896 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4896 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7752 0.1864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7752 0.1864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6538 -0.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6538 -0.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2041 1.8364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2041 1.8364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1438 0.1068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1438 0.1068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2261 3.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2261 3.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8509 1.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8509 1.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7752 -0.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7752 -0.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7972 0.7479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7972 0.7479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7752 2.5330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7752 2.5330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4818 2.9410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4818 2.9410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8109 2.7363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8109 2.7363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2071 -0.5587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2071 -0.5587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7945 -1.2734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7945 -1.2734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9962 -1.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9962 -1.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2027 -1.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2027 -1.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6153 -0.5763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6153 -0.5763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4137 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4137 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5820 -0.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5820 -0.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0620 0.1199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0620 0.1199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5548 -0.2110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5548 -0.2110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6599 -1.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6599 -1.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8265 -1.6976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8265 -1.6976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3277 -1.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3277 -1.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9151 -2.6431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9151 -2.6431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1167 -2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1167 -2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3232 -2.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3232 -2.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7358 -1.9460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7358 -1.9460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5342 -2.1551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5342 -2.1551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6912 -1.5693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6912 -1.5693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1710 -1.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1710 -1.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8109 -2.4116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8109 -2.4116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3637 -2.3841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3637 -2.3841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2882 -3.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2882 -3.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 1 23 1 0 0 0 0 | + | 1 23 1 0 0 0 0 |
| − | 13 24 1 0 0 0 0 | + | 13 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 27 35 1 0 0 0 0 | + | 27 35 1 0 0 0 0 |
| − | 28 36 1 0 0 0 0 | + | 28 36 1 0 0 0 0 |
| − | 29 37 1 0 0 0 0 | + | 29 37 1 0 0 0 0 |
| − | 30 22 1 0 0 0 0 | + | 30 22 1 0 0 0 0 |
| − | 38 39 1 1 0 0 0 | + | 38 39 1 1 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 41 40 1 1 0 0 0 | + | 41 40 1 1 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 38 1 0 0 0 0 | + | 43 38 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 38 46 1 0 0 0 0 | + | 38 46 1 0 0 0 0 |
| − | 39 47 1 0 0 0 0 | + | 39 47 1 0 0 0 0 |
| − | 40 48 1 0 0 0 0 | + | 40 48 1 0 0 0 0 |
| − | 41 37 1 0 0 0 0 | + | 41 37 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FFLGS0001 | + | ID FL5FFLGS0001 |
| − | KNApSAcK_ID C00013988 | + | KNApSAcK_ID C00013988 |
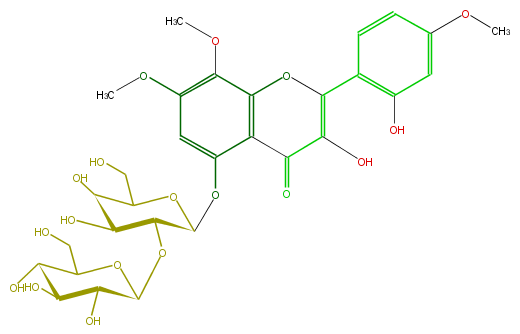
| − | NAME 3,5,2'-Trihydroxy-7,8,4'-trimethoxyflavone 5-glucosyl-(1->2)-galactoside;5-[(2-O-beta-D-Glucopyranosyl-beta-D-galactopyranosyl)oxy]-3-hydroxy-2-(2-hydroxy-4-methoxyphenyl)-7,8-dimethoxy-4H-1-benzopyran-4-one | + | NAME 3,5,2'-Trihydroxy-7,8,4'-trimethoxyflavone 5-glucosyl-(1->2)-galactoside;5-[(2-O-beta-D-Glucopyranosyl-beta-D-galactopyranosyl)oxy]-3-hydroxy-2-(2-hydroxy-4-methoxyphenyl)-7,8-dimethoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 527704-25-2 | + | CAS_RN 527704-25-2 |
| − | FORMULA C30H36O18 | + | FORMULA C30H36O18 |
| − | EXACTMASS 684.190164348 | + | EXACTMASS 684.190164348 |
| − | AVERAGEMASS 684.59604 | + | AVERAGEMASS 684.59604 |
| − | SMILES O(C1Oc(c35)cc(c(c3OC(=C(O)C5=O)c(c4O)ccc(OC)c4)OC)OC)C(CO)C(C(O)C1OC(O2)C(O)C(O)C(C2CO)O)O | + | SMILES O(C1Oc(c35)cc(c(c3OC(=C(O)C5=O)c(c4O)ccc(OC)c4)OC)OC)C(CO)C(C(O)C1OC(O2)C(O)C(O)C(C2CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
2.7972 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0827 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0827 2.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7972 3.0739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5117 2.6614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5117 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3682 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6538 1.8364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0607 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0607 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6538 0.1864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3682 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7752 1.8364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4896 1.4239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4896 0.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7752 0.1864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6538 -0.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2041 1.8364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1438 0.1068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2261 3.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8509 1.4629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7752 -0.5548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7972 0.7479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7752 2.5330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4818 2.9410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8109 2.7363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2071 -0.5587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7945 -1.2734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9962 -1.0642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2027 -1.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6153 -0.5763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4137 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5820 -0.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0620 0.1199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5548 -0.2110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6599 -1.0415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8265 -1.6976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3277 -1.9284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9151 -2.6431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1167 -2.4339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3232 -2.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7358 -1.9460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5342 -2.1551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6912 -1.5693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1710 -1.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8109 -2.4116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3637 -2.3841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2882 -3.0739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
18 21 1 0 0 0 0
16 22 1 0 0 0 0
1 23 1 0 0 0 0
13 24 1 0 0 0 0
24 25 1 0 0 0 0
20 26 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
27 35 1 0 0 0 0
28 36 1 0 0 0 0
29 37 1 0 0 0 0
30 22 1 0 0 0 0
38 39 1 1 0 0 0
39 40 1 1 0 0 0
41 40 1 1 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 38 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
38 46 1 0 0 0 0
39 47 1 0 0 0 0
40 48 1 0 0 0 0
41 37 1 0 0 0 0
S SKP 8
ID FL5FFLGS0001
KNApSAcK_ID C00013988
NAME 3,5,2'-Trihydroxy-7,8,4'-trimethoxyflavone 5-glucosyl-(1->2)-galactoside;5-[(2-O-beta-D-Glucopyranosyl-beta-D-galactopyranosyl)oxy]-3-hydroxy-2-(2-hydroxy-4-methoxyphenyl)-7,8-dimethoxy-4H-1-benzopyran-4-one
CAS_RN 527704-25-2
FORMULA C30H36O18
EXACTMASS 684.190164348
AVERAGEMASS 684.59604
SMILES O(C1Oc(c35)cc(c(c3OC(=C(O)C5=O)c(c4O)ccc(OC)c4)OC)OC)C(CO)C(C(O)C1OC(O2)C(O)C(O)C(C2CO)O)O
M END