Mol:FL5FAHGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2325 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2325 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2325 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2325 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6762 -0.1392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6762 -0.1392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1199 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1199 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1199 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1199 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6762 1.1455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6762 1.1455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5636 -0.1392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5636 -0.1392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0073 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0073 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0073 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0073 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5636 1.1455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5636 1.1455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5636 -0.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5636 -0.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1158 0.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1158 0.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6827 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6827 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6827 1.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6827 1.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1158 2.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1158 2.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 1.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 1.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6762 -0.7814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6762 -0.7814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 -0.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 -0.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4764 2.1794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4764 2.1794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8434 1.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8434 1.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2496 0.8181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2496 0.8181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7943 -1.7751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7943 -1.7751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4225 -2.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4225 -2.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2232 -1.4403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2232 -1.4403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4225 -0.7386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4225 -0.7386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7943 -0.3758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7943 -0.3758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9936 -1.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9936 -1.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7640 -1.0734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7640 -1.0734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9557 -2.3773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9557 -2.3773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2710 -2.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2710 -2.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8434 -1.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8434 -1.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3992 2.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3992 2.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1136 2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1136 2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 23 1 1 0 0 0 | + | 28 23 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | 16 33 1 0 0 0 0 | + | 16 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 33 34 | + | M SAL 1 2 33 34 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 36 -6.0287 5.6492 | + | M SBV 1 36 -6.0287 5.6492 |
| − | S SKP 8 | + | S SKP 8 |
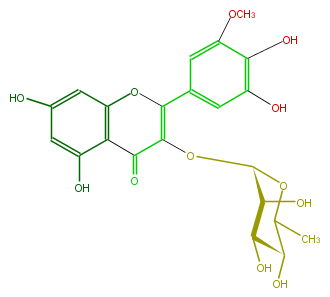
| − | ID FL5FAHGS0001 | + | ID FL5FAHGS0001 |
| − | KNApSAcK_ID C00005762 | + | KNApSAcK_ID C00005762 |
| − | NAME Laricitrin 3-rhamnoside | + | NAME Laricitrin 3-rhamnoside |
| − | CAS_RN 71367-37-8 | + | CAS_RN 71367-37-8 |
| − | FORMULA C22H22O12 | + | FORMULA C22H22O12 |
| − | EXACTMASS 478.111126168 | + | EXACTMASS 478.111126168 |
| − | AVERAGEMASS 478.40288000000004 | + | AVERAGEMASS 478.40288000000004 |
| − | SMILES OC(C1OC(C3=O)=C(c(c4)cc(OC)c(O)c(O)4)Oc(c32)cc(cc2O)O)C(C(O)C(C)O1)O | + | SMILES OC(C1OC(C3=O)=C(c(c4)cc(OC)c(O)c(O)4)Oc(c32)cc(cc2O)O)C(C(O)C(C)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-2.2325 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2325 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6762 -0.1392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1199 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1199 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6762 1.1455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5636 -0.1392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0073 0.1819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0073 0.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5636 1.1455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5636 -0.6401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1158 0.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6827 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6827 1.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1158 2.1274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 1.8000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6762 -0.7814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 -0.1391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4764 2.1794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8434 1.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2496 0.8181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7943 -1.7751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4225 -2.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2232 -1.4403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4225 -0.7386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7943 -0.3758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9936 -1.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7640 -1.0734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9557 -2.3773 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2710 -2.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8434 -1.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3992 2.7035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1136 2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
8 19 1 0 0 0 0
20 15 1 0 0 0 0
1 21 1 0 0 0 0
14 22 1 0 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 23 1 1 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
27 19 1 0 0 0 0
16 33 1 0 0 0 0
33 34 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 33 34
M SBL 1 1 36
M SMT 1 OCH3
M SBV 1 36 -6.0287 5.6492
S SKP 8
ID FL5FAHGS0001
KNApSAcK_ID C00005762
NAME Laricitrin 3-rhamnoside
CAS_RN 71367-37-8
FORMULA C22H22O12
EXACTMASS 478.111126168
AVERAGEMASS 478.40288000000004
SMILES OC(C1OC(C3=O)=C(c(c4)cc(OC)c(O)c(O)4)Oc(c32)cc(cc2O)O)C(C(O)C(C)O1)O
M END