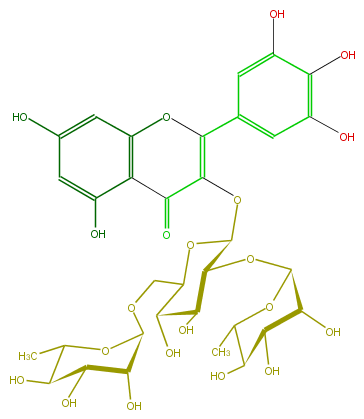
Mol:FL5FAGGS0025
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.8152 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8152 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1008 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1008 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1008 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1008 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8152 3.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8152 3.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5296 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5296 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5296 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5296 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3863 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3863 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3282 1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3282 1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0426 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0426 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0426 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0426 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3282 0.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3282 0.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3863 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3863 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7570 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7570 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4715 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4715 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4715 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4715 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7570 0.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7570 0.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3282 -0.5031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3282 -0.5031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1859 1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1859 1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1008 0.2039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1008 0.2039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7570 -0.4871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7570 -0.4871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2441 3.0913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2441 3.0913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8152 3.9163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8152 3.9163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2075 1.4625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2075 1.4625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5380 -2.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5380 -2.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2840 -2.0640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2840 -2.0640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4312 -1.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4312 -1.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9716 -0.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9716 -0.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1495 -0.7007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1495 -0.7007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0023 -1.5128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0023 -1.5128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3494 -0.9788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3494 -0.9788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2738 -2.8603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2738 -2.8603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0002 -2.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0002 -2.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5687 -1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5687 -1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0282 -1.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0282 -1.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3725 -2.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3725 -2.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6798 -3.5005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6798 -3.5005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9265 -3.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9265 -3.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1066 -3.2579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1066 -3.2579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7992 -2.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7992 -2.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5527 -2.8287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5527 -2.8287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2441 -3.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2441 -3.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9827 -2.9239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9827 -2.9239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3513 -3.8578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3513 -3.8578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0384 -3.9163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0384 -3.9163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0200 -2.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0200 -2.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2164 -3.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2164 -3.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6325 -2.4250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6325 -2.4250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3537 -2.0239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3537 -2.0239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1574 -1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1574 -1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7411 -1.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7411 -1.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9422 -2.4470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9422 -2.4470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7171 -3.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7171 -3.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7858 -3.3320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7858 -3.3320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6515 -2.8381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6515 -2.8381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 29 33 1 0 0 0 0 | + | 29 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 46 53 1 0 0 0 0 | + | 46 53 1 0 0 0 0 |
| − | 45 54 1 0 0 0 0 | + | 45 54 1 0 0 0 0 |
| − | 49 30 1 0 0 0 0 | + | 49 30 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0025 | + | ID FL5FAGGS0025 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES O(C(C(O)6)OC(C)C(O)C(O)6)CC(O2)C(C(C(C(OC(C(=O)3)=C(c(c5)cc(c(c5O)O)O)Oc(c4)c3c(O)cc4O)2)OC(O1)C(C(O)C(C1C)O)O)O)O | + | SMILES O(C(C(O)6)OC(C)C(O)C(O)6)CC(O2)C(C(C(C(OC(C(=O)3)=C(c(c5)cc(c(c5O)O)O)Oc(c4)c3c(O)cc4O)2)OC(O1)C(C(O)C(C1C)O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
1.8152 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1008 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1008 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8152 3.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5296 2.6788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5296 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3863 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3282 1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0426 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0426 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3282 0.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3863 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7570 1.8539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4715 1.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4715 0.6164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7570 0.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3282 -0.5031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1859 1.8539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1008 0.2039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7570 -0.4871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2441 3.0913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8152 3.9163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2075 1.4625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5380 -2.1365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2840 -2.0640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4312 -1.2519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9716 -0.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1495 -0.7007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0023 -1.5128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3494 -0.9788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2738 -2.8603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0002 -2.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5687 -1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0282 -1.9648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3725 -2.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6798 -3.5005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9265 -3.1638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1066 -3.2579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7992 -2.4919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5527 -2.8287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2441 -3.4070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9827 -2.9239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3513 -3.8578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0384 -3.9163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0200 -2.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2164 -3.1377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6325 -2.4250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3537 -2.0239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1574 -1.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7411 -1.9350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9422 -2.4470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7171 -3.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7858 -3.3320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6515 -2.8381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
26 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
29 33 1 0 0 0 0
33 34 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
34 39 1 0 0 0 0
27 19 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
47 52 1 0 0 0 0
46 53 1 0 0 0 0
45 54 1 0 0 0 0
49 30 1 0 0 0 0
S SKP 5
ID FL5FAGGS0025
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES O(C(C(O)6)OC(C)C(O)C(O)6)CC(O2)C(C(C(C(OC(C(=O)3)=C(c(c5)cc(c(c5O)O)O)Oc(c4)c3c(O)cc4O)2)OC(O1)C(C(O)C(C1C)O)O)O)O
M END