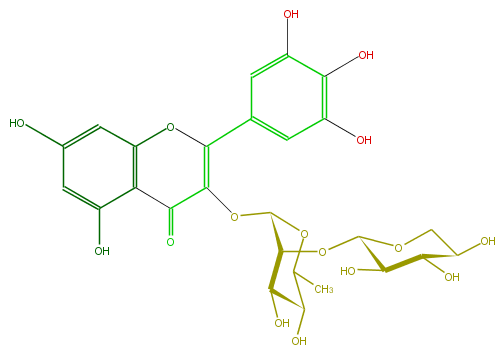
Mol:FL5FAGGS0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.7882 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7882 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7882 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7882 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0737 -0.6376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0737 -0.6376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3592 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3592 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3592 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3592 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0737 1.0123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0737 1.0123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6448 -0.6376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6448 -0.6376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9303 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9303 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9303 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9303 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6448 1.0123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6448 1.0123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6448 -1.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6448 -1.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0307 1.1712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0307 1.1712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6975 0.7507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6975 0.7507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4256 1.1712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4256 1.1712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4256 2.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4256 2.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6975 2.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6975 2.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0307 2.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0307 2.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0737 -1.4623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0737 -1.4623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2049 2.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2049 2.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6416 1.0926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6416 1.0926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3601 -0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3601 -0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6975 3.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6975 3.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1536 0.7508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1536 0.7508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3442 -2.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3442 -2.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0318 -2.6538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0318 -2.6538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8136 -1.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8136 -1.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0318 -1.1223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0318 -1.1223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3442 -0.7251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3442 -0.7251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5623 -1.4887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5623 -1.4887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4056 -1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4056 -1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5208 -2.9159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5208 -2.9159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8659 -3.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8659 -3.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3083 -2.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3083 -2.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1183 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1183 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6342 -1.8808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6342 -1.8808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3772 -1.5920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3772 -1.5920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0940 -1.5843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0940 -1.5843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5730 -1.0633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5730 -1.0633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9232 -1.4063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9232 -1.4063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9757 -1.8595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9757 -1.8595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9279 -1.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9279 -1.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6416 -1.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6416 -1.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 14 23 1 0 0 0 0 | + | 14 23 1 0 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 29 24 1 1 0 0 0 | + | 29 24 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 26 33 1 0 0 0 0 | + | 26 33 1 0 0 0 0 |
| − | 28 21 1 0 0 0 0 | + | 28 21 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0009 | + | ID FL5FAGGS0009 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES c(c13)(OC(=C(OC(C4OC(O5)C(C(C(C5)O)O)O)OC(C)C(C(O)4)O)C3=O)c(c2)cc(c(c(O)2)O)O)cc(cc1O)O | + | SMILES c(c13)(OC(=C(OC(C4OC(O5)C(C(C(C5)O)O)O)OC(C)C(C(O)4)O)C3=O)c(c2)cc(c(c(O)2)O)O)cc(cc1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.7882 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7882 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0737 -0.6376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3592 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3592 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0737 1.0123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6448 -0.6376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9303 -0.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9303 0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6448 1.0123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6448 -1.2808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0307 1.1712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6975 0.7507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4256 1.1712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4256 2.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6975 2.4324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0307 2.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0737 -1.4623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2049 2.4619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6416 1.0926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3601 -0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6975 3.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1536 0.7508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3442 -2.2568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0318 -2.6538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8136 -1.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0318 -1.1223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3442 -0.7251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5623 -1.4887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4056 -1.4887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5208 -2.9159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8659 -3.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3083 -2.2217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1183 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6342 -1.8808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3772 -1.5920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0940 -1.5843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5730 -1.0633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9232 -1.4063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9757 -1.8595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9279 -1.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6416 -1.2742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
16 22 1 0 0 0 0
14 23 1 0 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 1 0 0 0
29 24 1 1 0 0 0
29 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
26 33 1 0 0 0 0
28 21 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
35 40 1 0 0 0 0
36 41 1 0 0 0 0
37 42 1 0 0 0 0
30 34 1 0 0 0 0
S SKP 5
ID FL5FAGGS0009
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES c(c13)(OC(=C(OC(C4OC(O5)C(C(C(C5)O)O)O)OC(C)C(C(O)4)O)C3=O)c(c2)cc(c(c(O)2)O)O)cc(cc1O)O
M END