Mol:FL5FACGS0054
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.0026 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0026 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0026 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0026 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4463 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4463 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8900 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8900 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8900 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8900 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4463 0.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4463 0.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3337 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3337 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7774 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7774 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7774 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7774 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3337 0.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3337 0.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3337 -1.5965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3337 -1.5965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2213 0.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2213 0.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6543 -0.1384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6543 -0.1384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0874 0.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0874 0.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0874 0.8436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0874 0.8436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6543 1.1710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6543 1.1710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2213 0.8436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2213 0.8436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5587 0.1889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5587 0.1889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4795 1.1709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4795 1.1709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3832 -1.1634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3832 -1.1634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4463 -1.7378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4463 -1.7378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0055 -0.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0055 -0.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6062 -0.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6062 -0.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1055 -0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1055 -0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6062 0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6062 0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0978 0.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0978 0.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0978 1.3999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0978 1.3999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5587 1.6661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5587 1.6661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5587 2.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5587 2.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0978 2.4644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0978 2.4644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6369 2.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6369 2.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6369 1.6661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6369 1.6661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0978 2.9965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0978 2.9965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6543 1.8255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6543 1.8255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5945 -2.2053 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.5945 -2.2053 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.0591 -2.5144 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0591 -2.5144 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2290 -1.9199 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.2290 -1.9199 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0591 -1.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0591 -1.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5945 -1.0127 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5945 -1.0127 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.4247 -1.6072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.4247 -1.6072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2318 -1.6072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2318 -1.6072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9702 -2.5810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9702 -2.5810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0276 -3.0058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0276 -3.0058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2996 -2.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2996 -2.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1816 -1.2811 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.1816 -1.2811 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.5833 -1.8113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5833 -1.8113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1617 -1.5864 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1617 -1.5864 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7199 -1.5804 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7199 -1.5804 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3143 -1.1747 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.3143 -1.1747 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.8082 -1.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8082 -1.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9570 -1.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9570 -1.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4932 -2.1428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4932 -2.1428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1395 -0.9902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1395 -0.9902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7111 -1.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7111 -1.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 16 34 1 0 0 0 0 | + | 16 34 1 0 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 35 1 1 0 0 0 | + | 40 35 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 39 20 1 0 0 0 0 | + | 39 20 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 46 51 1 0 0 0 0 | + | 46 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 49 53 1 0 0 0 0 | + | 49 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 41 45 1 0 0 0 0 | + | 41 45 1 0 0 0 0 |
| − | 53 22 1 0 0 0 0 | + | 53 22 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0054 | + | ID FL5FACGS0054 |
| − | KNApSAcK_ID C00005978 | + | KNApSAcK_ID C00005978 |
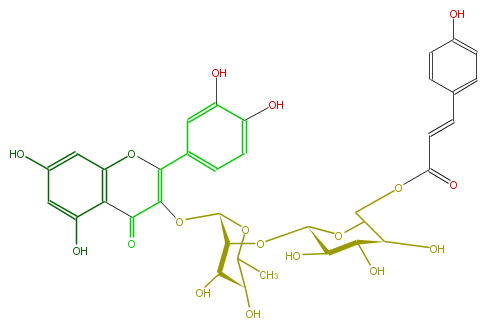
| − | NAME Quercetin 3-(6'''-p-coumarylglucosyl)(1->2)-rhamnoside | + | NAME Quercetin 3-(6'''-p-coumarylglucosyl)(1->2)-rhamnoside |
| − | CAS_RN 107190-71-6,143061-65-8 | + | CAS_RN 107190-71-6,143061-65-8 |
| − | FORMULA C36H36O18 | + | FORMULA C36H36O18 |
| − | EXACTMASS 756.190164348 | + | EXACTMASS 756.190164348 |
| − | AVERAGEMASS 756.6602399999999 | + | AVERAGEMASS 756.6602399999999 |
| − | SMILES [C@@H]([C@@H](OC(=C5c(c6)ccc(O)c6O)C(c(c(O5)4)c(O)cc(O)c4)=O)1)(O[C@H]([C@@H]3O)OC([C@H]([C@@H](O)3)O)COC(C=Cc(c2)ccc(c2)O)=O)[C@@H]([C@@H](C(C)O1)O)O | + | SMILES [C@@H]([C@@H](OC(=C5c(c6)ccc(O)c6O)C(c(c(O5)4)c(O)cc(O)c4)=O)1)(O[C@H]([C@@H]3O)OC([C@H]([C@@H](O)3)O)COC(C=Cc(c2)ccc(c2)O)=O)[C@@H]([C@@H](C(C)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-4.0026 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0026 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4463 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8900 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8900 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4463 0.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3337 -1.0957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7774 -0.7745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7774 -0.1321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3337 0.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3337 -1.5965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2213 0.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6543 -0.1384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0874 0.1889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0874 0.8436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6543 1.1710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2213 0.8436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5587 0.1889 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4795 1.1709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3832 -1.1634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4463 -1.7378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0055 -0.4902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6062 -0.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1055 -0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6062 0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0978 0.8334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0978 1.3999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5587 1.6661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5587 2.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0978 2.4644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6369 2.1983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6369 1.6661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0978 2.9965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6543 1.8255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5945 -2.2053 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.0591 -2.5144 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2290 -1.9199 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0591 -1.3219 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5945 -1.0127 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.4247 -1.6072 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2318 -1.6072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9702 -2.5810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0276 -3.0058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2996 -2.2251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1816 -1.2811 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.5833 -1.8113 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1617 -1.5864 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7199 -1.5804 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3143 -1.1747 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.8082 -1.4418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9570 -1.8418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4932 -2.1428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1395 -0.9902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7111 -1.7126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
23 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
32 27 1 0 0 0 0
30 33 1 0 0 0 0
16 34 1 0 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 1 0 0 0
40 35 1 1 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
36 43 1 0 0 0 0
37 44 1 0 0 0 0
39 20 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
46 51 1 0 0 0 0
47 52 1 0 0 0 0
49 53 1 0 0 0 0
48 54 1 0 0 0 0
41 45 1 0 0 0 0
53 22 1 0 0 0 0
S SKP 8
ID FL5FACGS0054
KNApSAcK_ID C00005978
NAME Quercetin 3-(6'''-p-coumarylglucosyl)(1->2)-rhamnoside
CAS_RN 107190-71-6,143061-65-8
FORMULA C36H36O18
EXACTMASS 756.190164348
AVERAGEMASS 756.6602399999999
SMILES [C@@H]([C@@H](OC(=C5c(c6)ccc(O)c6O)C(c(c(O5)4)c(O)cc(O)c4)=O)1)(O[C@H]([C@@H]3O)OC([C@H]([C@@H](O)3)O)COC(C=Cc(c2)ccc(c2)O)=O)[C@@H]([C@@H](C(C)O1)O)O
M END