Mol:FL5FACGL0069
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 70 76 0 0 0 0 0 0 0 0999 V2000 | + | 70 76 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.4248 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4248 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4248 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4248 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8685 0.4700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8685 0.4700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3122 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3122 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3122 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3122 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8685 1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8685 1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7559 0.4700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7559 0.4700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1996 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1996 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1996 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1996 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7559 1.7547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7559 1.7547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7559 -0.0308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7559 -0.0308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6435 1.7546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6435 1.7546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0765 1.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0765 1.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5095 1.7546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5095 1.7546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5095 2.4093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5095 2.4093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0765 2.7366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0765 2.7366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6435 2.4093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6435 2.4093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9809 1.7546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9809 1.7546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0573 2.7366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0573 2.7366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8054 0.4023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8054 0.4023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8685 -0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8685 -0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0765 3.3911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0765 3.3911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5473 -1.5954 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.5473 -1.5954 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.9651 -1.4451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.9651 -1.4451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.9929 -0.8504 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.9929 -0.8504 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7023 -0.3372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.7023 -0.3372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.3407 -0.5626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3407 -0.5626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2844 -1.1822 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2844 -1.1822 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.5473 -2.0682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5473 -2.0682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9651 -2.2015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9651 -2.2015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4596 -0.8432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4596 -0.8432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3596 -1.4641 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.3596 -1.4641 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.9985 -1.4641 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9985 -1.4641 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5393 -1.9083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5393 -1.9083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3823 -2.5313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.3823 -2.5313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2565 -2.5313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2565 -2.5313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2026 -2.0871 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.2026 -2.0871 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2558 -1.8733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2558 -1.8733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6218 -2.7016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6218 -2.7016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4654 -0.0262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4654 -0.0262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8600 0.2373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8600 0.2373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3695 -0.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3695 -0.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8600 0.7942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8600 0.7942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2659 1.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2659 1.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2659 1.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2659 1.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7663 1.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7663 1.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7663 2.5709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7663 2.5709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2659 2.8598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2659 2.8598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7655 2.5709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7655 2.5709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7655 1.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7655 1.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2659 3.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2659 3.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0862 -0.3342 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.0862 -0.3342 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.3155 -0.8644 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.3155 -0.8644 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8939 -0.6395 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8939 -0.6395 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.4108 -0.7159 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.4108 -0.7159 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0465 -0.2277 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.0465 -0.2277 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5404 -0.4948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5404 -0.4948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8499 -0.7751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8499 -0.7751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3107 -0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3107 -0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2254 -1.1959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2254 -1.1959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7690 -0.9228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7690 -0.9228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6843 -0.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6843 -0.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8920 -0.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8920 -0.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7923 -1.2048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7923 -1.2048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4083 3.1897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4083 3.1897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9084 4.0558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9084 4.0558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2077 -3.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2077 -3.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5068 -3.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5068 -3.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2664 2.8597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2664 2.8597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9809 2.4472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9809 2.4472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 33 31 1 0 0 0 0 | + | 33 31 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 52 53 1 1 0 0 0 | + | 52 53 1 1 0 0 0 |
| − | 53 54 1 1 0 0 0 | + | 53 54 1 1 0 0 0 |
| − | 55 54 1 1 0 0 0 | + | 55 54 1 1 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 52 1 0 0 0 0 | + | 57 52 1 0 0 0 0 |
| − | 52 58 1 0 0 0 0 | + | 52 58 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 58 32 1 0 0 0 0 | + | 58 32 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 62 40 1 0 0 0 0 | + | 62 40 1 0 0 0 0 |
| − | 28 63 1 0 0 0 0 | + | 28 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 49 65 1 0 0 0 0 | + | 49 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 35 67 1 0 0 0 0 | + | 35 67 1 0 0 0 0 |
| − | 67 68 1 0 0 0 0 | + | 67 68 1 0 0 0 0 |
| − | 47 69 1 0 0 0 0 | + | 47 69 1 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 67 68 | + | M SAL 4 2 67 68 |
| − | M SBL 4 1 73 | + | M SBL 4 1 73 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 73 -0.2077 -3.0248 | + | M SVB 4 73 -0.2077 -3.0248 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 63 64 | + | M SAL 3 2 63 64 |
| − | M SBL 3 1 69 | + | M SBL 3 1 69 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 69 -2.892 -0.7694 | + | M SVB 3 69 -2.892 -0.7694 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 69 70 | + | M SAL 2 2 69 70 |
| − | M SBL 2 1 75 | + | M SBL 2 1 75 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 75 4.2664 2.8597 | + | M SVB 2 75 4.2664 2.8597 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 65 66 | + | M SAL 1 2 65 66 |
| − | M SBL 1 1 71 | + | M SBL 1 1 71 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 71 2.4083 3.1897 | + | M SVB 1 71 2.4083 3.1897 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0069 | + | ID FL5FACGL0069 |
| − | KNApSAcK_ID C00005988 | + | KNApSAcK_ID C00005988 |
| − | NAME Quercetin 3-(6''''-sinapylsophorotrioside) | + | NAME Quercetin 3-(6''''-sinapylsophorotrioside) |
| − | CAS_RN 167638-67-7 | + | CAS_RN 167638-67-7 |
| − | FORMULA C44H50O26 | + | FORMULA C44H50O26 |
| − | EXACTMASS 994.2590317720001 | + | EXACTMASS 994.2590317720001 |
| − | AVERAGEMASS 994.8522 | + | AVERAGEMASS 994.8522 |
| − | SMILES COc(c1)c(c(cc(C=CC(=O)OCC([C@H]2O)O[C@H](OC(C7O)[C@@H](O[C@H](CO)[C@@H]7O)O[C@@H]([C@H]6O)[C@@H](OC([C@@H]6O)CO)OC(C4=O)=C(c(c5)ccc(c5O)O)Oc(c34)cc(O)cc(O)3)[C@@H]([C@@H]2O)O)1)OC)O | + | SMILES COc(c1)c(c(cc(C=CC(=O)OCC([C@H]2O)O[C@H](OC(C7O)[C@@H](O[C@H](CO)[C@@H]7O)O[C@@H]([C@H]6O)[C@@H](OC([C@@H]6O)CO)OC(C4=O)=C(c(c5)ccc(c5O)O)Oc(c34)cc(O)cc(O)3)[C@@H]([C@@H]2O)O)1)OC)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
70 76 0 0 0 0 0 0 0 0999 V2000
-4.4248 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4248 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8685 0.4700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3122 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3122 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8685 1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7559 0.4700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1996 0.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1996 1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7559 1.7547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7559 -0.0308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6435 1.7546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0765 1.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5095 1.7546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5095 2.4093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0765 2.7366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6435 2.4093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9809 1.7546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0573 2.7366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8054 0.4023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8685 -0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0765 3.3911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5473 -1.5954 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.9651 -1.4451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.9929 -0.8504 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7023 -0.3372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.3407 -0.5626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2844 -1.1822 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.5473 -2.0682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9651 -2.2015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4596 -0.8432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3596 -1.4641 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.9985 -1.4641 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5393 -1.9083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3823 -2.5313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2565 -2.5313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2026 -2.0871 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2558 -1.8733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6218 -2.7016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4654 -0.0262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8600 0.2373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3695 -0.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8600 0.7942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2659 1.0734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2659 1.7042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7663 1.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7663 2.5709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2659 2.8598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7655 2.5709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7655 1.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2659 3.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0862 -0.3342 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.3155 -0.8644 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8939 -0.6395 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.4108 -0.7159 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0465 -0.2277 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5404 -0.4948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8499 -0.7751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3107 -0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2254 -1.1959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7690 -0.9228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6843 -0.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8920 -0.7694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7923 -1.2048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4083 3.1897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9084 4.0558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2077 -3.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5068 -3.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2664 2.8597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9809 2.4472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 20 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
36 39 1 0 0 0 0
33 31 1 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
52 53 1 1 0 0 0
53 54 1 1 0 0 0
55 54 1 1 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
57 52 1 0 0 0 0
52 58 1 0 0 0 0
53 59 1 0 0 0 0
54 60 1 0 0 0 0
58 32 1 0 0 0 0
55 61 1 0 0 0 0
56 62 1 0 0 0 0
62 40 1 0 0 0 0
28 63 1 0 0 0 0
63 64 1 0 0 0 0
49 65 1 0 0 0 0
65 66 1 0 0 0 0
35 67 1 0 0 0 0
67 68 1 0 0 0 0
47 69 1 0 0 0 0
69 70 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 67 68
M SBL 4 1 73
M SMT 4 CH2OH
M SVB 4 73 -0.2077 -3.0248
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 63 64
M SBL 3 1 69
M SMT 3 CH2OH
M SVB 3 69 -2.892 -0.7694
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 69 70
M SBL 2 1 75
M SMT 2 OCH3
M SVB 2 75 4.2664 2.8597
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 65 66
M SBL 1 1 71
M SMT 1 OCH3
M SVB 1 71 2.4083 3.1897
S SKP 8
ID FL5FACGL0069
KNApSAcK_ID C00005988
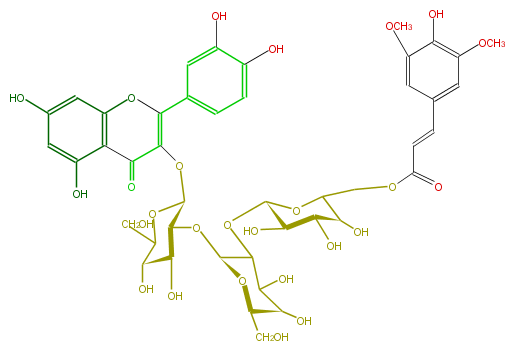
NAME Quercetin 3-(6''''-sinapylsophorotrioside)
CAS_RN 167638-67-7
FORMULA C44H50O26
EXACTMASS 994.2590317720001
AVERAGEMASS 994.8522
SMILES COc(c1)c(c(cc(C=CC(=O)OCC([C@H]2O)O[C@H](OC(C7O)[C@@H](O[C@H](CO)[C@@H]7O)O[C@@H]([C@H]6O)[C@@H](OC([C@@H]6O)CO)OC(C4=O)=C(c(c5)ccc(c5O)O)Oc(c34)cc(O)cc(O)3)[C@@H]([C@@H]2O)O)1)OC)O
M END