Mol:FL5FAAGS0073
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.6034 1.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6034 1.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6034 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6034 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3179 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3179 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0324 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0324 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0324 1.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0324 1.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3179 1.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3179 1.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8890 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8890 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1745 0.5654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1745 0.5654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4600 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4600 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4600 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4600 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1745 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1745 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8890 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8890 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7456 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7456 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0311 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0311 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0311 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0311 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7456 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7456 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1745 -1.8029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1745 -1.8029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6834 0.5654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6834 0.5654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4953 -1.0222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4953 -1.0222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6331 1.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6331 1.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7456 -1.7842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7456 -1.7842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3845 0.8592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3845 0.8592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9720 0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9720 0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1740 0.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1740 0.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3807 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3807 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7932 0.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7932 0.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5913 0.6326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5913 0.6326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7482 1.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7482 1.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2279 1.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2279 1.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8676 0.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8676 0.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4206 0.4037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4206 0.4037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3454 -0.2859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3454 -0.2859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1500 -0.2793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1500 -0.2793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7375 -0.9937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7375 -0.9937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9395 -0.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9395 -0.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1462 -1.0114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1462 -1.0114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5587 -0.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5587 -0.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3568 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3568 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5137 0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5137 0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9934 0.3567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9934 0.3567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6331 -0.7623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6331 -0.7623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1861 -0.7348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1861 -0.7348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1109 -1.4244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1109 -1.4244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGS0073 | + | ID FL5FAAGS0073 |
| − | KNApSAcK_ID C00013740 | + | KNApSAcK_ID C00013740 |
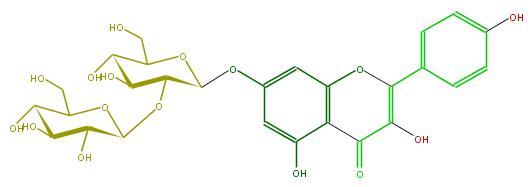
| − | NAME Kaempferol 7-sophoroside;7-[(2-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-3,5-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Kaempferol 7-sophoroside;7-[(2-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-3,5-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 195621-97-7 | + | CAS_RN 195621-97-7 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES Oc(c5)ccc(c5)C(=C(O)4)Oc(c1C4=O)cc(OC(O3)C(C(O)C(C(CO)3)O)OC(O2)C(C(O)C(C2CO)O)O)cc1O | + | SMILES Oc(c5)ccc(c5)C(=C(O)4)Oc(c1C4=O)cc(OC(O3)C(C(O)C(C(CO)3)O)OC(O2)C(C(O)C(C2CO)O)O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
3.6034 1.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6034 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3179 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0324 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0324 1.3904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3179 1.8029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8890 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1745 0.5654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4600 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4600 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1745 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8890 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7456 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0311 0.1529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0311 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7456 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1745 -1.8029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6834 0.5654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4953 -1.0222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6331 1.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7456 -1.7842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3845 0.8592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9720 0.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1740 0.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3807 0.1271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7932 0.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5913 0.6326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7482 1.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2279 1.4952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8676 0.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4206 0.4037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3454 -0.2859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1500 -0.2793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7375 -0.9937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9395 -0.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1462 -1.0114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5587 -0.2969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3568 -0.5059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5137 0.0797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9934 0.3567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6331 -0.7623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1861 -0.7348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1109 -1.4244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 32 1 0 0 0 0
25 18 1 0 0 0 0
S SKP 8
ID FL5FAAGS0073
KNApSAcK_ID C00013740
NAME Kaempferol 7-sophoroside;7-[(2-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-3,5-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 195621-97-7
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES Oc(c5)ccc(c5)C(=C(O)4)Oc(c1C4=O)cc(OC(O3)C(C(O)C(C(CO)3)O)OC(O2)C(C(O)C(C2CO)O)O)cc1O
M END