Mol:FL5FAAGL0071
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2010 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2010 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2010 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2010 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6447 0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6447 0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0884 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0884 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0884 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0884 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6447 2.2684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6447 2.2684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5321 0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5321 0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9758 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9758 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9758 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9758 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5321 2.2684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5321 2.2684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5321 0.4829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5321 0.4829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4197 2.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4197 2.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1472 1.9410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1472 1.9410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7142 2.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7142 2.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7142 2.9230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7142 2.9230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1472 3.2504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1472 3.2504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4197 2.9230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4197 2.9230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3277 0.7546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3277 0.7546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7177 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7177 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3301 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3301 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0755 0.7876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0755 0.7876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8255 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8255 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2132 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2132 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4677 1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4677 1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0094 1.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0094 1.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7365 1.4986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7365 1.4986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6995 1.1157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6995 1.1157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2810 3.2503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2810 3.2503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6447 0.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6447 0.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8120 2.1458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8120 2.1458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4544 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4544 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8000 -0.0240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8000 -0.0240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7596 -0.7959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7596 -0.7959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4247 -0.7959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4247 -0.7959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1408 -1.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1408 -1.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1408 -1.9096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1408 -1.9096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5093 -2.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5093 -2.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9457 -1.9488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9457 -1.9488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3822 -2.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3822 -2.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3822 -2.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3822 -2.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9457 -3.2504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9457 -3.2504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5093 -2.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5093 -2.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1811 -3.2502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1811 -3.2502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1927 1.5348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1927 1.5348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8120 1.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8120 1.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1927 2.1566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1927 2.1566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 27 44 1 0 0 0 0 | + | 27 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0071 | + | ID FL5FAAGL0071 |
| − | KNApSAcK_ID C00005853 | + | KNApSAcK_ID C00005853 |
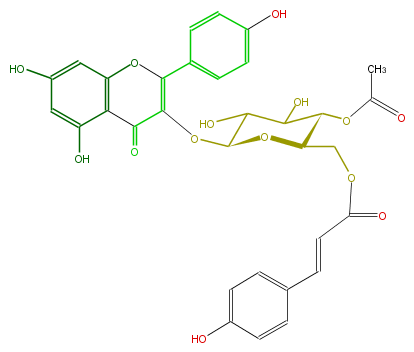
| − | NAME Kaempferol 3-(4''-acetyl-6''-p-coumarylglucoside) | + | NAME Kaempferol 3-(4''-acetyl-6''-p-coumarylglucoside) |
| − | CAS_RN 115651-95-1 | + | CAS_RN 115651-95-1 |
| − | FORMULA C32H28O14 | + | FORMULA C32H28O14 |
| − | EXACTMASS 636.147905604 | + | EXACTMASS 636.147905604 |
| − | AVERAGEMASS 636.55632 | + | AVERAGEMASS 636.55632 |
| − | SMILES c(O)(c1)ccc(C(=C(OC(O4)C(O)C(C(OC(C)=O)C4COC(=O)C=Cc(c5)ccc(O)c5)O)3)Oc(c(C3=O)2)cc(cc(O)2)O)c1 | + | SMILES c(O)(c1)ccc(C(=C(OC(O4)C(O)C(C(OC(C)=O)C4COC(=O)C=Cc(c5)ccc(O)c5)O)3)Oc(c(C3=O)2)cc(cc(O)2)O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-3.2010 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2010 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6447 0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0884 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0884 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6447 2.2684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5321 0.9837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9758 1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9758 1.9473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5321 2.2684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5321 0.4829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4197 2.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1472 1.9410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7142 2.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7142 2.9230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1472 3.2504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4197 2.9230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3277 0.7546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7177 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3301 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0755 0.7876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8255 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2132 1.2460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4677 1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0094 1.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7365 1.4986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6995 1.1157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2810 3.2503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6447 0.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8120 2.1458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4544 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8000 -0.0240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7596 -0.7959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4247 -0.7959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1408 -1.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1408 -1.9096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5093 -2.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9457 -1.9488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3822 -2.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3822 -2.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9457 -3.2504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5093 -2.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1811 -3.2502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1927 1.5348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8120 1.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1927 2.1566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
22 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
27 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
S SKP 8
ID FL5FAAGL0071
KNApSAcK_ID C00005853
NAME Kaempferol 3-(4''-acetyl-6''-p-coumarylglucoside)
CAS_RN 115651-95-1
FORMULA C32H28O14
EXACTMASS 636.147905604
AVERAGEMASS 636.55632
SMILES c(O)(c1)ccc(C(=C(OC(O4)C(O)C(C(OC(C)=O)C4COC(=O)C=Cc(c5)ccc(O)c5)O)3)Oc(c(C3=O)2)cc(cc(O)2)O)c1
M END