Mol:FL3FAIGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5242 -0.6589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5242 -0.6589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5242 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5242 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8415 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8415 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1589 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1589 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1589 -0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1589 -0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8415 -0.3271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8415 -0.3271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5239 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5239 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2067 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2067 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2067 -0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2067 -0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5239 -0.3271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5239 -0.3271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5239 -2.7293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5239 -2.7293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8891 -0.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8891 -0.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5849 -0.7291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5849 -0.7291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2806 -0.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2806 -0.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2806 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2806 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5849 0.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5849 0.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8891 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8891 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8415 -2.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8415 -2.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1499 0.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1499 0.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2887 -0.2531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2887 -0.2531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0374 -0.1862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0374 -0.1862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5171 -0.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5171 -0.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7679 -0.5816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7679 -0.5816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0450 -0.5738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0450 -0.5738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5703 -0.0484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5703 -0.0484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3356 -0.3231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3356 -0.3231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0195 0.1837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0195 0.1837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2855 -0.7694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2855 -0.7694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1014 -1.0126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1014 -1.0126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4384 -1.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4384 -1.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8295 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8295 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0773 -2.1618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0773 -2.1618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3206 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3206 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9294 -1.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9294 -1.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6816 -1.9142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6816 -1.9142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2880 -1.7733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2880 -1.7733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4069 -2.1729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4069 -2.1729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6416 -2.7651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6416 -2.7651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3605 -3.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3605 -3.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0960 -0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0960 -0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4069 -1.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4069 -1.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5849 1.7053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5849 1.7053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1727 3.1001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1727 3.1001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6916 0.4818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6916 0.4818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6916 1.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6916 1.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1129 1.3083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1129 1.3083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 14 40 1 0 0 0 0 | + | 14 40 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 26 44 1 0 0 0 0 | + | 26 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 45 -0.8154 0.4708 | + | M SBV 1 45 -0.8154 0.4708 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 47 0.0000 -0.8275 | + | M SBV 2 47 0.0000 -0.8275 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 3 44 45 46 | + | M SAL 3 3 44 45 46 |
| − | M SBL 3 1 50 | + | M SBL 3 1 50 |
| − | M SMT 3 ^ COOH | + | M SMT 3 ^ COOH |
| − | M SBV 3 50 0.3560 -0.8049 | + | M SBV 3 50 0.3560 -0.8049 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAIGS0005 | + | ID FL3FAIGS0005 |
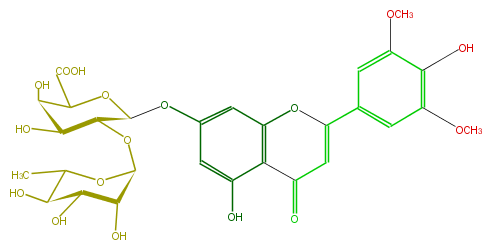
| − | FORMULA C29H32O17 | + | FORMULA C29H32O17 |
| − | EXACTMASS 652.163949598 | + | EXACTMASS 652.163949598 |
| − | AVERAGEMASS 652.55418 | + | AVERAGEMASS 652.55418 |
| − | SMILES c(C(O2)=CC(=O)c(c5O)c2cc(c5)OC(C(OC(C(O)4)OC(C)C(C4O)O)3)OC(C(O)C3O)C(O)=O)(c1)cc(OC)c(c(OC)1)O | + | SMILES c(C(O2)=CC(=O)c(c5O)c2cc(c5)OC(C(OC(C(O)4)OC(C)C(C4O)O)3)OC(C(O)C3O)C(O)=O)(c1)cc(OC)c(c(OC)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-1.5242 -0.6589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5242 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8415 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1589 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1589 -0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8415 -0.3271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5239 -1.9038 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2067 -1.5096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2067 -0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5239 -0.3271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5239 -2.7293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8891 -0.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5849 -0.7291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2806 -0.3273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2806 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5849 0.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8891 0.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8415 -2.6905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1499 0.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2887 -0.2531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0374 -0.1862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5171 -0.8729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7679 -0.5816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0450 -0.5738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5703 -0.0484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3356 -0.3231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0195 0.1837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2855 -0.7694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1014 -1.0126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4384 -1.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8295 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0773 -2.1618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3206 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9294 -1.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6816 -1.9142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2880 -1.7733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4069 -2.1729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6416 -2.7651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3605 -3.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0960 -0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4069 -1.5547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5849 1.7053 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1727 3.1001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6916 0.4818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6916 1.4908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1129 1.3083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
20 1 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 29 1 0 0 0 0
24 20 1 0 0 0 0
40 41 1 0 0 0 0
14 40 1 0 0 0 0
42 43 1 0 0 0 0
16 42 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
26 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 OCH3
M SBV 1 45 -0.8154 0.4708
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 47
M SMT 2 OCH3
M SBV 2 47 0.0000 -0.8275
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 3 44 45 46
M SBL 3 1 50
M SMT 3 ^ COOH
M SBV 3 50 0.3560 -0.8049
S SKP 5
ID FL3FAIGS0005
FORMULA C29H32O17
EXACTMASS 652.163949598
AVERAGEMASS 652.55418
SMILES c(C(O2)=CC(=O)c(c5O)c2cc(c5)OC(C(OC(C(O)4)OC(C)C(C4O)O)3)OC(C(O)C3O)C(O)=O)(c1)cc(OC)c(c(OC)1)O
M END