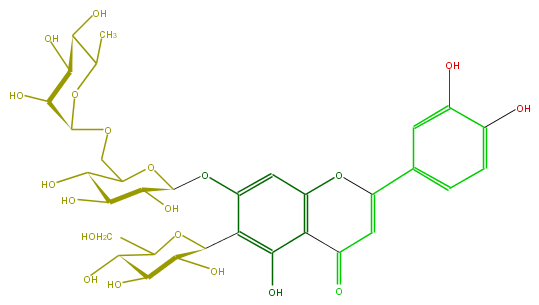
Mol:FL3FACDS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6982 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6982 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6982 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6982 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0485 -2.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0485 -2.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7952 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7952 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7952 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7952 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0485 -0.5159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0485 -0.5159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5419 -2.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5419 -2.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2885 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2885 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2885 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2885 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5419 -0.5159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5419 -0.5159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5419 -3.0814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5419 -3.0814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4446 -0.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4446 -0.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0485 -3.1023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0485 -3.1023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2011 -0.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2011 -0.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9879 -0.8320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9879 -0.8320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7748 -0.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7748 -0.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7748 0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7748 0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9879 0.9851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9879 0.9851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2011 0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2011 0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5612 0.9848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5612 0.9848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9879 1.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9879 1.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3660 -2.3033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3660 -2.3033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7145 -2.8095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7145 -2.8095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0938 -2.3543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0938 -2.3543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4269 -2.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4269 -2.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0431 -1.8121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0431 -1.8121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5636 -2.2946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5636 -2.2946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0787 -2.8232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0787 -2.8232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3868 -2.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3868 -2.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2485 -2.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2485 -2.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0628 -0.4729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0628 -0.4729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5646 -1.1304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5646 -1.1304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8472 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8472 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1550 -0.8440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1550 -0.8440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6579 -0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6579 -0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2857 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2857 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8546 -0.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8546 -0.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3953 -1.0611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3953 -1.0611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2698 -1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2698 -1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7552 -0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7552 -0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5965 0.4981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5965 0.4981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9278 1.1049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9278 1.1049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4045 0.4813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4045 0.4813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3470 1.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3470 1.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8675 1.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8675 1.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3907 2.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3907 2.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4483 1.7684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4483 1.7684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8604 3.1023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8604 3.1023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7371 2.6210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7371 2.6210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9433 2.5355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9433 2.5355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5612 1.2811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5612 1.2811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4349 -1.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4349 -1.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1641 -2.2908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1641 -2.2908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 9 14 1 0 0 0 0 | + | 9 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 18 1 0 0 0 0 | + | 21 18 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 2 1 0 0 0 0 | + | 25 2 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 34 12 1 0 0 0 0 | + | 34 12 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 42 1 1 0 0 0 | + | 47 42 1 1 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 42 51 1 0 0 0 0 | + | 42 51 1 0 0 0 0 |
| − | 43 41 1 0 0 0 0 | + | 43 41 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 27 52 1 0 0 0 0 | + | 27 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 58 0.8713 -0.4352 | + | M SBV 1 58 0.8713 -0.4352 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACDS0013 | + | ID FL3FACDS0013 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES c(c(C(=C6)Oc(c3)c(C6=O)c(c(c(OC(C5O)OC(C(C(O)5)O)COC(C4O)OC(C(O)C(O)4)C)3)C(O2)C(C(C(C2CO)O)O)O)O)1)cc(O)c(c1)O | + | SMILES c(c(C(=C6)Oc(c3)c(C6=O)c(c(c(OC(C5O)OC(C(C(O)5)O)COC(C4O)OC(C(O)C(O)4)C)3)C(O2)C(C(C(C2CO)O)O)O)O)1)cc(O)c(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-0.6982 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6982 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0485 -2.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7952 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7952 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0485 -0.5159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5419 -2.2404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2885 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2885 -0.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5419 -0.5159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5419 -3.0814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4446 -0.5162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0485 -3.1023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2011 -0.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9879 -0.8320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7748 -0.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7748 0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9879 0.9851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2011 0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5612 0.9848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9879 1.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3660 -2.3033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7145 -2.8095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0938 -2.3543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4269 -2.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0431 -1.8121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5636 -2.2946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0787 -2.8232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3868 -2.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2485 -2.6478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0628 -0.4729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5646 -1.1304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8472 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1550 -0.8440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6579 -0.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2857 -0.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8546 -0.7141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3953 -1.0611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2698 -1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7552 -0.1811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5965 0.4981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9278 1.1049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4045 0.4813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3470 1.2934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8675 1.9567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3907 2.5803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4483 1.7684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8604 3.1023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7371 2.6210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9433 2.5355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5612 1.2811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4349 -1.8593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1641 -2.2908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
9 14 1 0 0 0 0
17 20 1 0 0 0 0
21 18 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 2 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
36 40 1 0 0 0 0
34 12 1 0 0 0 0
40 41 1 0 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 42 1 1 0 0 0
46 48 1 0 0 0 0
45 49 1 0 0 0 0
47 50 1 0 0 0 0
42 51 1 0 0 0 0
43 41 1 0 0 0 0
52 53 1 0 0 0 0
27 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 ^ CH2OH
M SBV 1 58 0.8713 -0.4352
S SKP 5
ID FL3FACDS0013
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES c(c(C(=C6)Oc(c3)c(C6=O)c(c(c(OC(C5O)OC(C(C(O)5)O)COC(C4O)OC(C(O)C(O)4)C)3)C(O2)C(C(C(C2CO)O)O)O)O)1)cc(O)c(c1)O
M END