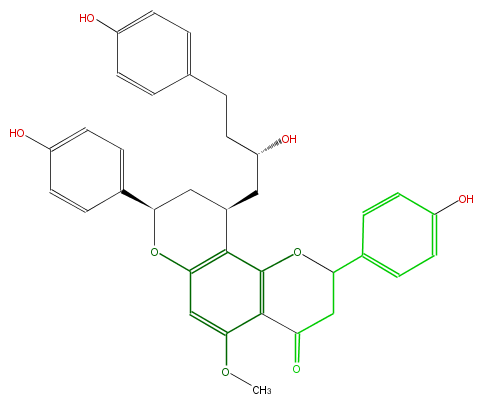
Mol:FL2FBANC0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 48 0 0 0 0 0 0 0 0999 V2000 | + | 43 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2824 -2.6620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2824 -2.6620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0011 -2.2570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0011 -2.2570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4280 -2.2426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4280 -2.2426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4192 -1.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4192 -1.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2995 -1.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2995 -1.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0097 -1.4320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0097 -1.4320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3081 -0.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3081 -0.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0268 0.2179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0268 0.2179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7369 -0.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7369 -0.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7284 -1.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7284 -1.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3959 0.1699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3959 0.1699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4039 0.9948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4039 0.9948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1224 1.4003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1224 1.4003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8328 0.9809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8328 0.9809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8247 0.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8247 0.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1062 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1062 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4293 1.3175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4293 1.3175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2751 -3.4081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2751 -3.4081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3356 -3.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3356 -3.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1422 -2.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1422 -2.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8542 -2.2331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8542 -2.2331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8471 -1.4107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8471 -1.4107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1313 -0.9998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1313 -0.9998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1467 -3.3365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1467 -3.3365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3114 0.1759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3114 0.1759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3064 0.9318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3064 0.9318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8620 1.2575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8620 1.2575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3225 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3225 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3278 2.0955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3278 2.0955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0790 2.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0790 2.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0844 3.3475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0844 3.3475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8016 3.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8016 3.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5133 3.3381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5133 3.3381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5079 2.5131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5079 2.5131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7907 2.1053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7907 2.1053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0454 3.6406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0454 3.6406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4355 -1.0658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4355 -1.0658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1527 -1.4735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1527 -1.4735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8644 -1.0563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8644 -1.0563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8589 -0.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8589 -0.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1418 0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1418 0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4300 -0.2408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4300 -0.2408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4293 0.1030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4293 0.1030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 1 0 0 0 | + | 9 11 1 1 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 4 1 0 0 0 0 | + | 23 4 1 0 0 0 0 |
| − | 20 24 2 0 0 0 0 | + | 20 24 2 0 0 0 0 |
| − | 7 25 1 1 0 0 0 | + | 7 25 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 6 0 0 0 | + | 26 27 1 6 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 22 37 1 0 0 0 0 | + | 22 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FBANC0006 | + | ID FL2FBANC0006 |
| − | KNApSAcK_ID C00014290 | + | KNApSAcK_ID C00014290 |
| − | NAME Calyxin M | + | NAME Calyxin M |
| − | CAS_RN 332877-84-6 | + | CAS_RN 332877-84-6 |
| − | FORMULA C35H34O8 | + | FORMULA C35H34O8 |
| − | EXACTMASS 582.225368064 | + | EXACTMASS 582.225368064 |
| − | AVERAGEMASS 582.63966 | + | AVERAGEMASS 582.63966 |
| − | SMILES O(c52)C(CC(CC(O)CCc(c6)ccc(c6)O)c2c(c(c(c5)OC)4)OC(CC(=O)4)c(c3)ccc(c3)O)c(c1)ccc(O)c1 | + | SMILES O(c52)C(CC(CC(O)CCc(c6)ccc(c6)O)c2c(c(c(c5)OC)4)OC(CC(=O)4)c(c3)ccc(c3)O)c(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 48 0 0 0 0 0 0 0 0999 V2000
-0.2824 -2.6620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0011 -2.2570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4280 -2.2426 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4192 -1.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2995 -1.0121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0097 -1.4320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3081 -0.1872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0268 0.2179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7369 -0.2020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7284 -1.0269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3959 0.1699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4039 0.9948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1224 1.4003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8328 0.9809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8247 0.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1062 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4293 1.3175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2751 -3.4081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3356 -3.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1422 -2.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8542 -2.2331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8471 -1.4107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1313 -0.9998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1467 -3.3365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3114 0.1759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3064 0.9318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8620 1.2575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3225 1.2893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3278 2.0955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0790 2.5226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0844 3.3475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8016 3.7553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5133 3.3381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5079 2.5131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7907 2.1053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0454 3.6406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4355 -1.0658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1527 -1.4735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8644 -1.0563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8589 -0.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1418 0.1764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4300 -0.2408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4293 0.1030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 1 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
18 19 1 0 0 0 0
3 20 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 4 1 0 0 0 0
20 24 2 0 0 0 0
7 25 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 6 0 0 0
26 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 30 1 0 0 0 0
33 36 1 0 0 0 0
22 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
S SKP 8
ID FL2FBANC0006
KNApSAcK_ID C00014290
NAME Calyxin M
CAS_RN 332877-84-6
FORMULA C35H34O8
EXACTMASS 582.225368064
AVERAGEMASS 582.63966
SMILES O(c52)C(CC(CC(O)CCc(c6)ccc(c6)O)c2c(c(c(c5)OC)4)OC(CC(=O)4)c(c3)ccc(c3)O)c(c1)ccc(O)c1
M END