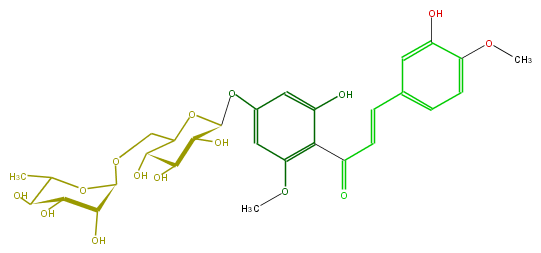
Mol:FL1CBEGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 47 0 0 0 0 0 0 0 0999 V2000 | + | 44 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2642 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2642 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2642 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2642 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4503 -0.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4503 -0.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1648 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1648 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1648 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1648 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4503 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4503 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8792 -0.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8792 -0.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5937 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5937 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5937 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5937 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3082 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3082 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0226 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0226 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7371 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7371 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7371 1.6464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7371 1.6464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0226 2.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0226 2.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3082 1.6464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3082 1.6464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8792 -1.6536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8792 -1.6536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8638 0.8363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8638 0.8363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4183 2.0397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4183 2.0397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8319 0.7941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8319 0.7941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1003 1.6460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1003 1.6460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0226 2.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0226 2.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4503 -1.5727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4503 -1.5727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2659 -1.9862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2659 -1.9862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9508 -0.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9508 -0.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2229 -0.9566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2229 -0.9566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7967 -0.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7967 -0.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1049 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1049 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8329 0.3116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8329 0.3116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2591 -0.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2591 -0.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2058 -0.3730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2058 -0.3730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1807 -1.1762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1807 -1.1762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6745 -1.2369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6745 -1.2369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8138 -0.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8138 -0.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6878 -0.8294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6878 -0.8294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2655 -1.2344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2655 -1.2344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7611 -1.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7611 -1.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9461 -1.7646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9461 -1.7646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1361 -2.0490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1361 -2.0490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6831 -1.4085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6831 -1.4085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4982 -1.5381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4982 -1.5381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1003 -1.6619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1003 -1.6619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9480 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9480 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4318 -2.1047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4318 -2.1047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2023 -2.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2023 -2.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 13 18 1 0 0 0 0 | + | 13 18 1 0 0 0 0 |
| − | 5 19 1 0 0 0 0 | + | 5 19 1 0 0 0 0 |
| − | 18 20 1 0 0 0 0 | + | 18 20 1 0 0 0 0 |
| − | 14 21 1 0 0 0 0 | + | 14 21 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 29 33 1 0 0 0 0 | + | 29 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 27 17 1 0 0 0 0 | + | 27 17 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1CBEGS0001 | + | ID FL1CBEGS0001 |
| − | FORMULA C29H36O15 | + | FORMULA C29H36O15 |
| − | EXACTMASS 624.2054204819999 | + | EXACTMASS 624.2054204819999 |
| − | AVERAGEMASS 624.58714 | + | AVERAGEMASS 624.58714 |
| − | SMILES c(C(=O)C=Cc(c4)cc(O)c(c4)OC)(c1OC)c(cc(OC(O2)C(O)C(O)C(O)C2COC(O3)C(C(C(O)C(C)3)O)O)c1)O | + | SMILES c(C(=O)C=Cc(c4)cc(O)c(c4)OC)(c1OC)c(cc(OC(O2)C(O)C(O)C(O)C2COC(O3)C(C(C(O)C(C)3)O)O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 47 0 0 0 0 0 0 0 0999 V2000
-0.2642 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2642 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4503 -0.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1648 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1648 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4503 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8792 -0.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5937 -0.4161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5937 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3082 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0226 0.4089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7371 0.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7371 1.6464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0226 2.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3082 1.6464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8792 -1.6536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8638 0.8363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4183 2.0397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8319 0.7941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1003 1.6460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0226 2.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4503 -1.5727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2659 -1.9862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9508 -0.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2229 -0.9566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7967 -0.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1049 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8329 0.3116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2591 -0.3431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2058 -0.3730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1807 -1.1762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6745 -1.2369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8138 -0.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6878 -0.8294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2655 -1.2344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7611 -1.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9461 -1.7646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1361 -2.0490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6831 -1.4085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4982 -1.5381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1003 -1.6619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9480 -1.1888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4318 -2.1047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2023 -2.7688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
1 17 1 0 0 0 0
13 18 1 0 0 0 0
5 19 1 0 0 0 0
18 20 1 0 0 0 0
14 21 1 0 0 0 0
3 22 1 0 0 0 0
22 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
26 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
29 33 1 0 0 0 0
33 34 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
34 39 1 0 0 0 0
27 17 1 0 0 0 0
S SKP 5
ID FL1CBEGS0001
FORMULA C29H36O15
EXACTMASS 624.2054204819999
AVERAGEMASS 624.58714
SMILES c(C(=O)C=Cc(c4)cc(O)c(c4)OC)(c1OC)c(cc(OC(O2)C(O)C(O)C(O)C2COC(O3)C(C(C(O)C(C)3)O)O)c1)O
M END