Mol:FL5FEGNS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 28 30 0 0 0 0 0 0 0 0999 V2000 | + | 28 30 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2602 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2602 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8164 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8164 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8165 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8165 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2601 -1.9489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2601 -1.9489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7039 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7039 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7039 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7039 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2602 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2602 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7038 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7038 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1476 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1476 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1476 -1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1476 -1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6939 -2.8417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6939 -2.8417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4085 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4085 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4085 -3.5671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4085 -3.5671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9755 -3.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9755 -3.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5425 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5425 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5425 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5425 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9755 -2.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9755 -2.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2602 -0.0221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2602 -0.0221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3725 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3725 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0425 -4.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0425 -4.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1831 -2.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1831 -2.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1809 -2.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1809 -2.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9749 -0.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9749 -0.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1180 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1180 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7038 -3.9124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7038 -3.9124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7038 -4.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7038 -4.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9755 -4.8943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9755 -4.8943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9755 -5.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9755 -5.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 8 25 1 0 0 0 0 | + | 8 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 14 27 1 0 0 0 0 | + | 14 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 27 28 | + | M SAL 4 2 27 28 |
| − | M SBL 4 1 29 | + | M SBL 4 1 29 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 29 2.0624 -0.022 | + | M SVB 4 29 2.0624 -0.022 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 25 26 | + | M SAL 3 2 25 26 |
| − | M SBL 3 1 27 | + | M SBL 3 1 27 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 27 0.3617 -0.9854 | + | M SVB 3 27 0.3617 -0.9854 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 23 24 | + | M SAL 2 2 23 24 |
| − | M SBL 2 1 25 | + | M SBL 2 1 25 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 25 -2.7769 -0.6877 | + | M SVB 2 25 -2.7769 -0.6877 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 21 22 | + | M SAL 1 2 21 22 |
| − | M SBL 1 1 23 | + | M SBL 1 1 23 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 23 1.5693 1.7424 | + | M SVB 1 23 1.5693 1.7424 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FEGNS0008 | + | ID FL5FEGNS0008 |
| − | KNApSAcK_ID C00004828 | + | KNApSAcK_ID C00004828 |
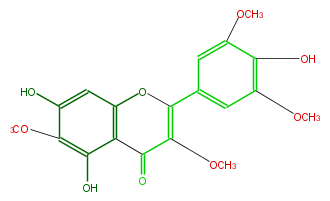
| − | NAME 5,7,4'-Trihydroxy-3,6,3',5'-tetramethoxyflavone | + | NAME 5,7,4'-Trihydroxy-3,6,3',5'-tetramethoxyflavone |
| − | CAS_RN 63296-16-2 | + | CAS_RN 63296-16-2 |
| − | FORMULA C19H18O9 | + | FORMULA C19H18O9 |
| − | EXACTMASS 390.095082174 | + | EXACTMASS 390.095082174 |
| − | AVERAGEMASS 390.34082 | + | AVERAGEMASS 390.34082 |
| − | SMILES c(c3OC)(O)cc(O1)c(c3O)C(C(=C1c(c2)cc(c(c(OC)2)O)OC)OC)=O | + | SMILES c(c3OC)(O)cc(O1)c(c3O)C(C(=C1c(c2)cc(c(c(OC)2)O)OC)OC)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
28 30 0 0 0 0 0 0 0 0999 V2000
-1.2602 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8164 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8165 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2601 -1.9489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7039 -1.6277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7039 -0.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2602 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7038 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1476 -2.5913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1476 -1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6939 -2.8417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4085 -2.9123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4085 -3.5671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9755 -3.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5425 -3.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5425 -2.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9755 -2.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2602 -0.0221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3725 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0425 -4.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1831 -2.8698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1809 -2.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9749 -0.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1180 -1.0952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7038 -3.9124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7038 -4.9124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9755 -4.8943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9755 -5.8943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
21 22 1 0 0 0 0
2 23 1 0 0 0 0
23 24 1 0 0 0 0
8 25 1 0 0 0 0
25 26 1 0 0 0 0
14 27 1 0 0 0 0
27 28 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 27 28
M SBL 4 1 29
M SMT 4 OCH3
M SVB 4 29 2.0624 -0.022
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 25 26
M SBL 3 1 27
M SMT 3 OCH3
M SVB 3 27 0.3617 -0.9854
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 23 24
M SBL 2 1 25
M SMT 2 OCH3
M SVB 2 25 -2.7769 -0.6877
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 21 22
M SBL 1 1 23
M SMT 1 OCH3
M SVB 1 23 1.5693 1.7424
S SKP 8
ID FL5FEGNS0008
KNApSAcK_ID C00004828
NAME 5,7,4'-Trihydroxy-3,6,3',5'-tetramethoxyflavone
CAS_RN 63296-16-2
FORMULA C19H18O9
EXACTMASS 390.095082174
AVERAGEMASS 390.34082
SMILES c(c3OC)(O)cc(O1)c(c3O)C(C(=C1c(c2)cc(c(c(OC)2)O)OC)OC)=O
M END