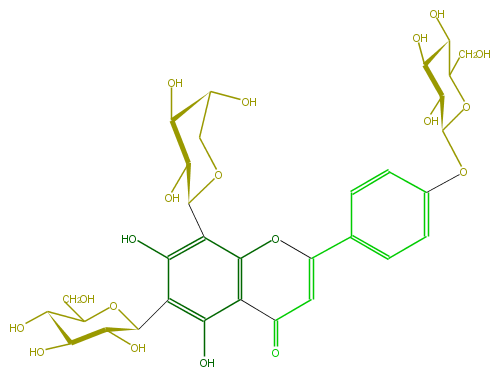
Mol:FL3FAADS0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7039 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7039 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7039 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7039 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9894 -2.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9894 -2.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2750 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2750 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2750 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2750 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9894 -1.1704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9894 -1.1704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4395 -2.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4395 -2.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1540 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1540 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1540 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1540 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4395 -1.1704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4395 -1.1704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4395 -3.4636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4395 -3.4636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4181 -1.1706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4181 -1.1706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8599 1.7616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8599 1.7616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6377 1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6377 1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3077 0.3240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3077 0.3240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2989 -0.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2989 -0.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7038 0.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7038 0.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0955 0.8425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0955 0.8425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1624 1.5748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1624 1.5748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6377 1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6377 1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7024 -0.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7024 -0.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9894 -3.6451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9894 -3.6451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9477 -1.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9477 -1.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7005 -1.5787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7005 -1.5787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4534 -1.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4534 -1.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4534 -0.2748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4534 -0.2748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7005 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7005 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9477 -0.2748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9477 -0.2748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2057 0.1597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2057 0.1597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1328 -2.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1328 -2.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6562 -3.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6562 -3.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9697 -3.0085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9697 -3.0085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3072 -3.0015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3072 -3.0015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7886 -2.5200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7886 -2.5200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3891 -2.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3891 -2.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6552 -2.9479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6552 -2.9479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2648 -3.4386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2648 -3.4386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3752 -3.3518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3752 -3.3518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9134 2.7838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9134 2.7838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4153 2.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4153 2.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7641 1.6738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7641 1.6738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7641 0.9693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7641 0.9693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2623 1.4674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2623 1.4674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9134 2.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9134 2.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5988 3.3288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5988 3.3288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2654 2.8449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2654 2.8449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4862 1.1925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4862 1.1925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8571 -2.3691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8571 -2.3691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8173 -1.9211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8173 -1.9211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1752 2.5328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1752 2.5328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8173 3.6451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8173 3.6451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 9 1 0 0 0 0 | + | 23 9 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 2 1 0 0 0 0 | + | 33 2 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 39 45 1 0 0 0 0 | + | 39 45 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 29 1 0 0 0 0 | + | 42 29 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 35 48 1 0 0 0 0 | + | 35 48 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 48 49 | + | M SAL 1 2 48 49 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 54 0.4679 -0.4679 | + | M SBV 1 54 0.4679 -0.4679 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 50 51 | + | M SAL 2 2 50 51 |
| − | M SBL 2 1 56 | + | M SBL 2 1 56 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 56 -0.2617 -0.4533 | + | M SBV 2 56 -0.2617 -0.4533 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAADS0021 | + | ID FL3FAADS0021 |
| − | FORMULA C32H38O19 | + | FORMULA C32H38O19 |
| − | EXACTMASS 726.200729034 | + | EXACTMASS 726.200729034 |
| − | AVERAGEMASS 726.6327200000001 | + | AVERAGEMASS 726.6327200000001 |
| − | SMILES c(c5O)(C(O6)C(C(O)C(O)C6CO)O)c(O)c(c(c54)OC(=CC(=O)4)c(c3)ccc(c3)OC(O2)C(O)C(C(O)C(CO)2)O)C(O1)C(O)C(C(C1)O)O | + | SMILES c(c5O)(C(O6)C(C(O)C(O)C6CO)O)c(O)c(c(c54)OC(=CC(=O)4)c(c3)ccc(c3)OC(O2)C(O)C(C(O)C(CO)2)O)C(O1)C(O)C(C(C1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
-1.7039 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7039 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9894 -2.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2750 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2750 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9894 -1.1704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4395 -2.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1540 -2.4079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1540 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4395 -1.1704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4395 -3.4636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4181 -1.1706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8599 1.7616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6377 1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3077 0.3240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2989 -0.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7038 0.1001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0955 0.8425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1624 1.5748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6377 1.9489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7024 -0.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9894 -3.6451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9477 -1.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7005 -1.5787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4534 -1.1441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4534 -0.2748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7005 0.1600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9477 -0.2748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2057 0.1597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1328 -2.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6562 -3.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9697 -3.0085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3072 -3.0015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7886 -2.5200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3891 -2.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6552 -2.9479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2648 -3.4386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3752 -3.3518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9134 2.7838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4153 2.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7641 1.6738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7641 0.9693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2623 1.4674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9134 2.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5988 3.3288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2654 2.8449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4862 1.1925 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8571 -2.3691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8173 -1.9211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1752 2.5328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8173 3.6451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
23 9 1 0 0 0 0
26 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 2 1 0 0 0 0
39 40 1 1 0 0 0
40 41 1 1 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 39 1 0 0 0 0
39 45 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 29 1 0 0 0 0
48 49 1 0 0 0 0
35 48 1 0 0 0 0
50 51 1 0 0 0 0
44 50 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 48 49
M SBL 1 1 54
M SMT 1 ^ CH2OH
M SBV 1 54 0.4679 -0.4679
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 50 51
M SBL 2 1 56
M SMT 2 CH2OH
M SBV 2 56 -0.2617 -0.4533
S SKP 5
ID FL3FAADS0021
FORMULA C32H38O19
EXACTMASS 726.200729034
AVERAGEMASS 726.6327200000001
SMILES c(c5O)(C(O6)C(C(O)C(O)C6CO)O)c(O)c(c(c54)OC(=CC(=O)4)c(c3)ccc(c3)OC(O2)C(O)C(C(O)C(CO)2)O)C(O1)C(O)C(C(C1)O)O
M END