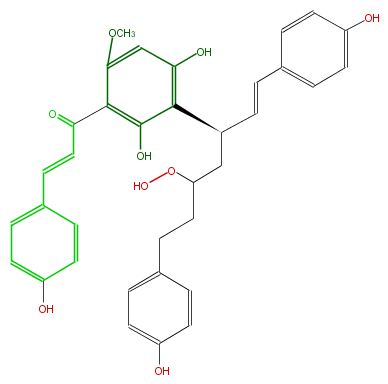
Mol:FL1CBANC0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 47 0 0 0 0 0 0 0 0999 V2000 | + | 44 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.6744 0.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6744 0.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3442 0.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3442 0.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3442 1.4549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3442 1.4549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6744 1.8416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6744 1.8416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0046 1.4549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0046 1.4549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0046 0.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0046 0.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6720 1.8455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6720 1.8455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1947 1.5437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1947 1.5437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7923 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7923 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3727 1.5536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3727 1.5536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0205 1.9276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0205 1.9276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6683 1.5536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6683 1.5536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6683 0.8056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6683 0.8056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0205 0.4316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0205 0.4316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3727 0.8056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3727 0.8056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1492 0.5280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1492 0.5280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6720 2.3079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6720 2.3079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5282 0.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5282 0.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8916 0.3654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8916 0.3654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6744 -0.8107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.6744 -0.8107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3561 -1.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3561 -1.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8829 -0.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8829 -0.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3999 -1.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3999 -1.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3999 -1.9381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3999 -1.9381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0404 -2.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0404 -2.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6808 -1.9381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6808 -1.9381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6808 -1.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6808 -1.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0404 -0.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0404 -0.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1492 -2.2085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1492 -2.2085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1076 -1.1379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1076 -1.1379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4617 -0.8092 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.4617 -0.8092 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.0984 -1.1768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0984 -1.1768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7809 -0.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7809 -0.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3767 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3767 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9859 -0.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9859 -0.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5950 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5950 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5950 -1.8301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5950 -1.8301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9859 -2.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9859 -2.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3767 -1.8301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3767 -1.8301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0537 -2.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0537 -2.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4617 -0.3153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4617 -0.3153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0066 -0.0007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0066 -0.0007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3317 2.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3317 2.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2727 2.4164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2727 2.4164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 13 16 1 0 0 0 0 | + | 13 16 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 2 19 1 0 0 0 0 | + | 2 19 1 0 0 0 0 |
| − | 20 1 1 1 0 0 0 | + | 20 1 1 1 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 2 0 0 0 0 | + | 21 22 2 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 20 30 1 0 0 0 0 | + | 20 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 31 41 1 0 0 0 0 | + | 31 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 4 43 1 0 0 0 0 | + | 4 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 46 1.3317 2.078 | + | M SVB 1 46 1.3317 2.078 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CBANC0001 | + | ID FL1CBANC0001 |
| − | KNApSAcK_ID C00008123 | + | KNApSAcK_ID C00008123 |
| − | NAME Calyxin A | + | NAME Calyxin A |
| − | CAS_RN 164991-52-0 | + | CAS_RN 164991-52-0 |
| − | FORMULA C35H34O9 | + | FORMULA C35H34O9 |
| − | EXACTMASS 598.220282686 | + | EXACTMASS 598.220282686 |
| − | AVERAGEMASS 598.63906 | + | AVERAGEMASS 598.63906 |
| − | SMILES c(c2O)c(OC)c(c(c([C@@H](C=Cc(c4)ccc(c4)O)CC(OO)CCc(c3)ccc(c3)O)2)O)C(C=Cc(c1)ccc(c1)O)=O | + | SMILES c(c2O)c(OC)c(c(c([C@@H](C=Cc(c4)ccc(c4)O)CC(OO)CCc(c3)ccc(c3)O)2)O)C(C=Cc(c1)ccc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 47 0 0 0 0 0 0 0 0999 V2000
0.6744 0.2948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3442 0.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3442 1.4549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6744 1.8416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0046 1.4549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0046 0.6815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6720 1.8455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1947 1.5437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7923 1.8887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3727 1.5536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0205 1.9276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6683 1.5536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6683 0.8056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0205 0.4316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3727 0.8056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1492 0.5280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6720 2.3079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5282 0.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8916 0.3654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6744 -0.8107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3561 -1.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8829 -0.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3999 -1.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3999 -1.9381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0404 -2.3079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6808 -1.9381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6808 -1.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0404 -0.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1492 -2.2085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1076 -1.1379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4617 -0.8092 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.0984 -1.1768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7809 -0.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3767 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9859 -0.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5950 -1.1267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5950 -1.8301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9859 -2.1818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3767 -1.8301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0537 -2.0949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4617 -0.3153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0066 -0.0007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3317 2.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2727 2.4164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
13 16 1 0 0 0 0
7 17 2 0 0 0 0
6 18 1 0 0 0 0
2 19 1 0 0 0 0
20 1 1 1 0 0 0
20 21 1 0 0 0 0
21 22 2 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
26 29 1 0 0 0 0
20 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
31 41 1 0 0 0 0
41 42 1 0 0 0 0
4 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 46
M SMT 1 OCH3
M SVB 1 46 1.3317 2.078
S SKP 8
ID FL1CBANC0001
KNApSAcK_ID C00008123
NAME Calyxin A
CAS_RN 164991-52-0
FORMULA C35H34O9
EXACTMASS 598.220282686
AVERAGEMASS 598.63906
SMILES c(c2O)c(OC)c(c(c([C@@H](C=Cc(c4)ccc(c4)O)CC(OO)CCc(c3)ccc(c3)O)2)O)C(C=Cc(c1)ccc(c1)O)=O
M END