Mol:FL5FAGGS0031
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.7862 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7862 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 2.2822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 2.2822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7862 2.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7862 2.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 2.2822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 2.2822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 1.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 1.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0717 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0717 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0717 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0717 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 -0.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 -0.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7862 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7862 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5006 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5006 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5006 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5006 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7862 -0.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7862 -0.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 -0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 -0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2151 1.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2151 1.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 -0.1928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 -0.1928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7862 -0.8838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7862 -0.8838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2151 2.6947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2151 2.6947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7862 3.5197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7862 3.5197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1786 1.0658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1786 1.0658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0366 -1.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0366 -1.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1025 -2.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1025 -2.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5631 -1.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5631 -1.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3844 -1.6263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3844 -1.6263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5236 -0.8129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5236 -0.8129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8580 -1.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8580 -1.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7201 -2.2942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7201 -2.2942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3558 -2.4945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3558 -2.4945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5821 -2.2479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5821 -2.2479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5411 -1.6871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5411 -1.6871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7157 -3.1020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7157 -3.1020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0361 -3.4600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0361 -3.4600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3871 -3.5197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3871 -3.5197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 24 33 1 0 0 0 0 | + | 24 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 28 19 1 0 0 0 0 | + | 28 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0031 | + | ID FL5FAGGS0031 |
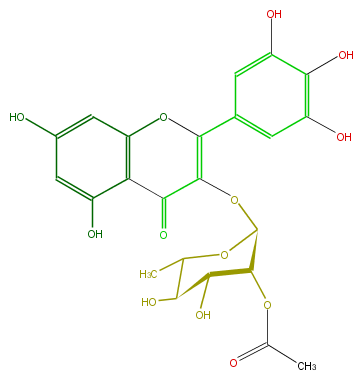
| − | FORMULA C23H22O13 | + | FORMULA C23H22O13 |
| − | EXACTMASS 506.10604078999995 | + | EXACTMASS 506.10604078999995 |
| − | AVERAGEMASS 506.41298000000006 | + | AVERAGEMASS 506.41298000000006 |
| − | SMILES C(C)(C(O)1)OC(OC(C4=O)=C(Oc(c43)cc(cc3O)O)c(c2)cc(O)c(O)c(O)2)C(OC(C)=O)C(O)1 | + | SMILES C(C)(C(O)1)OC(OC(C4=O)=C(Oc(c43)cc(cc3O)O)c(c2)cc(O)c(O)c(O)2)C(OC(C)=O)C(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
1.7862 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 2.2822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7862 2.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 2.2822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 1.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0717 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0717 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 -0.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7862 1.4572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5006 1.0447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5006 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7862 -0.1928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 -0.8998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2151 1.4572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 -0.1928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7862 -0.8838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2151 2.6947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7862 3.5197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1786 1.0658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0366 -1.3815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1025 -2.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5631 -1.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3844 -1.6263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5236 -0.8129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8580 -1.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7201 -2.2942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3558 -2.4945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5821 -2.2479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5411 -1.6871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7157 -3.1020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0361 -3.4600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3871 -3.5197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
27 30 1 0 0 0 0
26 31 1 0 0 0 0
25 32 1 0 0 0 0
24 33 1 0 0 0 0
30 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
28 19 1 0 0 0 0
S SKP 5
ID FL5FAGGS0031
FORMULA C23H22O13
EXACTMASS 506.10604078999995
AVERAGEMASS 506.41298000000006
SMILES C(C)(C(O)1)OC(OC(C4=O)=C(Oc(c43)cc(cc3O)O)c(c2)cc(O)c(O)c(O)2)C(OC(C)=O)C(O)1
M END