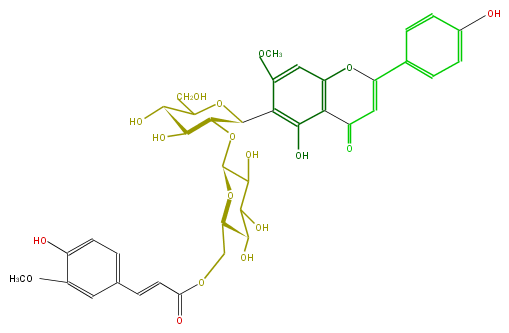
Mol:FL3FCACS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.9358 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9358 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9358 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9358 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6503 1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6503 1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3648 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3648 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3648 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3648 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6503 2.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6503 2.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0793 1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0793 1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7938 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7938 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7938 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7938 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0793 2.8203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0793 2.8203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0793 0.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0793 0.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6503 0.3454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6503 0.3454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6671 2.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6671 2.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4200 2.5178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4200 2.5178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1729 2.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1729 2.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1729 3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1729 3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4200 4.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4200 4.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6671 3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6671 3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0271 4.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0271 4.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1551 1.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1551 1.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4929 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4929 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8306 1.3309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8306 1.3309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0701 1.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0701 1.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5656 1.8077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5656 1.8077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2809 1.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2809 1.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8125 1.2957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8125 1.2957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1722 0.8262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1722 0.8262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2242 0.8667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2242 0.8667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2282 -0.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2282 -0.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4824 -0.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4824 -0.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2568 -0.7986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2568 -0.7986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4824 -1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4824 -1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2282 -2.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2282 -2.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0027 -1.2136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0027 -1.2136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2317 0.3614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2317 0.3614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5071 -1.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5071 -1.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0934 -2.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0934 -2.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4384 -2.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4384 -2.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0696 -3.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0696 -3.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6906 -3.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6906 -3.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2741 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2741 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8567 -3.5994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8567 -3.5994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4395 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4395 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6906 -4.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6906 -4.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4395 -2.4560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4395 -2.4560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1384 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1384 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8373 -2.4560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8373 -2.4560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8373 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8373 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1384 -3.6665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1384 -3.6665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5347 -2.0534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5347 -2.0534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7549 -3.1359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7549 -3.1359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.0271 -2.9595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.0271 -2.9595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8408 2.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8408 2.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0248 3.0481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0248 3.0481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4770 3.2025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4770 3.2025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1651 4.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1651 4.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 2 1 0 0 0 0 | + | 23 2 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 30 28 1 0 0 0 0 | + | 30 28 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 40 44 2 0 0 0 0 | + | 40 44 2 0 0 0 0 |
| − | 43 45 2 0 0 0 0 | + | 43 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 43 1 0 0 0 0 | + | 49 43 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 25 53 1 0 0 0 0 | + | 25 53 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 1 55 1 0 0 0 0 | + | 1 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 57 0.9176 -0.1272 | + | M SBV 1 57 0.9176 -0.1272 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 59 | + | M SBL 2 1 59 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 59 0.5598 -0.5414 | + | M SBV 2 59 0.5598 -0.5414 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 61 | + | M SBL 3 1 61 |
| − | M SMT 3 ^ OCH3 | + | M SMT 3 ^ OCH3 |
| − | M SBV 3 61 0.4588 -0.7948 | + | M SBV 3 61 0.4588 -0.7948 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCACS0024 | + | ID FL3FCACS0024 |
| − | FORMULA C38H40O18 | + | FORMULA C38H40O18 |
| − | EXACTMASS 784.2214644759999 | + | EXACTMASS 784.2214644759999 |
| − | AVERAGEMASS 784.7134 | + | AVERAGEMASS 784.7134 |
| − | SMILES C(C(=O)OCC(O2)C(O)C(O)C(O)C(OC(C3c(c6O)c(cc(c65)OC(=CC5=O)c(c4)ccc(c4)O)OC)C(C(O)C(CO)O3)O)2)=Cc(c1)ccc(c(OC)1)O | + | SMILES C(C(=O)OCC(O2)C(O)C(O)C(O)C(OC(C3c(c6O)c(cc(c65)OC(=CC5=O)c(c4)ccc(c4)O)OC)C(C(O)C(CO)O3)O)2)=Cc(c1)ccc(c(OC)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
0.9358 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9358 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6503 1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3648 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3648 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6503 2.8203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0793 1.1702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7938 1.5826 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7938 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0793 2.8203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0793 0.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6503 0.3454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6671 2.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4200 2.5178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1729 2.9526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1729 3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4200 4.2566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6671 3.8220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0271 4.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1551 1.6753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4929 0.9335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8306 1.3309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0701 1.2249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5656 1.8077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2809 1.4633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8125 1.2957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1722 0.8262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2242 0.8667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2282 -0.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4824 -0.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2568 -0.7986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4824 -1.5922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2282 -2.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0027 -1.2136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2317 0.3614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5071 -1.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0934 -2.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4384 -2.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0696 -3.2414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6906 -3.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2741 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8567 -3.5994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4395 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6906 -4.3152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4395 -2.4560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1384 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8373 -2.4560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8373 -3.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1384 -3.6665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5347 -2.0534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7549 -3.1359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.0271 -2.9595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8408 2.0047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0248 3.0481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4770 3.2025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1651 4.3149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 2 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
30 28 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
40 44 2 0 0 0 0
43 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 43 1 0 0 0 0
47 50 1 0 0 0 0
51 52 1 0 0 0 0
48 51 1 0 0 0 0
53 54 1 0 0 0 0
25 53 1 0 0 0 0
55 56 1 0 0 0 0
1 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 ^ OCH3
M SBV 1 57 0.9176 -0.1272
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 59
M SMT 2 ^ CH2OH
M SBV 2 59 0.5598 -0.5414
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 61
M SMT 3 ^ OCH3
M SBV 3 61 0.4588 -0.7948
S SKP 5
ID FL3FCACS0024
FORMULA C38H40O18
EXACTMASS 784.2214644759999
AVERAGEMASS 784.7134
SMILES C(C(=O)OCC(O2)C(O)C(O)C(O)C(OC(C3c(c6O)c(cc(c65)OC(=CC5=O)c(c4)ccc(c4)O)OC)C(C(O)C(CO)O3)O)2)=Cc(c1)ccc(c(OC)1)O
M END