Mol:BMCCPUAPf028
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 46 0 0 1 0 0 0 0 0999 V2000 | + | 44 46 0 0 1 0 0 0 0 0999 V2000 |
| − | 3.7321 2.8496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 2.8496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 1.3496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 1.3496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 0.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 0.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 1.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 1.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1229 0.6805 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1229 0.6805 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5296 -0.2330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5296 -0.2330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5241 -0.1285 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5241 -0.1285 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 2.8496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 2.8496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1933 -0.8717 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.1933 -0.8717 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.9854 -1.8498 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.9854 -1.8498 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.8514 -2.3498 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.8514 -2.3498 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.5945 -1.6807 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.5945 -1.6807 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 6.5727 -1.8886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5727 -1.8886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0718 -2.2565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0718 -2.2565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9559 -3.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9559 -3.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1878 -0.7671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1878 -0.7671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2418 -1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2418 -1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2200 -1.3534 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2200 -1.3534 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0120 -2.3315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0120 -2.3315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4279 -0.3752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4279 -0.3752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1981 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1981 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8672 -0.8181 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8672 -0.8181 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6104 -1.4873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6104 -1.4873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1241 -0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1241 -0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5364 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5364 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5145 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5145 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1836 0.4603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1836 0.4603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9268 -0.2089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9268 -0.2089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4405 1.1294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4405 1.1294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.8528 1.2034 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 12.8528 1.2034 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 13.8309 0.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8309 0.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5000 1.7386 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5000 1.7386 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.4782 1.5307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.4782 1.5307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1473 2.2739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1473 2.2739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1255 2.0660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1255 2.0660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.7946 2.8091 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.7946 2.8091 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.7728 2.6012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.7728 2.6012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.4419 3.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.4419 3.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.4200 3.1364 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.4200 3.1364 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5438 2.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5438 2.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.1399 0.0444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.1399 0.0444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.4345 1.1149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.4345 1.1149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 6 2 0 0 0 0 | + | 1 6 2 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 5 4 2 0 0 0 0 | + | 5 4 2 0 0 0 0 |
| − | 9 8 1 0 0 0 0 | + | 9 8 1 0 0 0 0 |
| − | 8 7 2 0 0 0 0 | + | 8 7 2 0 0 0 0 |
| − | 7 5 1 0 0 0 0 | + | 7 5 1 0 0 0 0 |
| − | 4 9 1 0 0 0 0 | + | 4 9 1 0 0 0 0 |
| − | 6 10 1 0 0 0 0 | + | 6 10 1 0 0 0 0 |
| − | 14 18 1 6 0 0 0 | + | 14 18 1 6 0 0 0 |
| − | 14 13 1 0 0 0 0 | + | 14 13 1 0 0 0 0 |
| − | 11 18 1 6 0 0 0 | + | 11 18 1 6 0 0 0 |
| − | 13 12 1 0 0 0 0 | + | 13 12 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 11 9 1 0 0 0 0 | + | 11 9 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 20 22 2 0 0 0 0 | + | 20 22 2 0 0 0 0 |
| − | 20 23 1 0 0 0 0 | + | 20 23 1 0 0 0 0 |
| − | 27 24 1 0 0 0 0 | + | 27 24 1 0 0 0 0 |
| − | 24 26 2 0 0 0 0 | + | 24 26 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 24 23 1 0 0 0 0 | + | 24 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 29 31 1 0 0 0 0 | + | 29 31 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 32 42 1 1 0 0 0 | + | 32 42 1 1 0 0 0 |
| − | 33 43 2 0 0 0 0 | + | 33 43 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 37 44 2 0 0 0 0 | + | 37 44 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 12 16 1 1 0 0 0 | + | 12 16 1 1 0 0 0 |
| − | 13 17 1 1 0 0 0 | + | 13 17 1 1 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPUAPf028 | + | ID BMCCPUAPf028 |
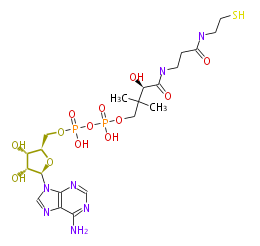
| − | NAME Dephospho-CoA | + | NAME Dephospho-CoA |
| − | FORMULA C21H35N7O13P2S | + | FORMULA C21H35N7O13P2S |
| − | EXACTMASS 687.1488 | + | EXACTMASS 687.1488 |
| − | AVERAGEMASS 687.5555 | + | AVERAGEMASS 687.5555 |
| − | SMILES P(OP(OC[C@H]([C@H]3O)O[C@H]([C@@H]3O)n(c2)c(n1)c(n2)c(N)nc1)(O)=O)(OCC(C)(C)[C@H](C(=O)NCCC(=O)NCCS)O)(O)=O | + | SMILES P(OP(OC[C@H]([C@H]3O)O[C@H]([C@@H]3O)n(c2)c(n1)c(n2)c(N)nc1)(O)=O)(OCC(C)(C)[C@H](C(=O)NCCC(=O)NCCS)O)(O)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00882 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00882 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 46 0 0 1 0 0 0 0 0999 V2000
3.7321 2.8496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 1.3496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 0.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 1.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 2.3496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1229 0.6805 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.5296 -0.2330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5241 -0.1285 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 2.8496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.1933 -0.8717 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.9854 -1.8498 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.8514 -2.3498 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.5945 -1.6807 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
6.5727 -1.8886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0718 -2.2565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9559 -3.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1878 -0.7671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.2418 -1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.2200 -1.3534 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
8.0120 -2.3315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.4279 -0.3752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.1981 -1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.8672 -0.8181 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
10.6104 -1.4873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.1241 -0.1490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.5364 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.5145 -0.2829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.1836 0.4603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.9268 -0.2089 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.4405 1.1294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.8528 1.2034 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
13.8309 0.9955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.5000 1.7386 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
15.4782 1.5307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.1473 2.2739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.1255 2.0660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.7946 2.8091 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
18.7728 2.6012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
19.4419 3.3443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
20.4200 3.1364 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
12.5438 2.1545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.1399 0.0444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.4345 1.1149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 6 2 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
5 6 1 0 0 0 0
5 4 2 0 0 0 0
9 8 1 0 0 0 0
8 7 2 0 0 0 0
7 5 1 0 0 0 0
4 9 1 0 0 0 0
6 10 1 0 0 0 0
14 18 1 6 0 0 0
14 13 1 0 0 0 0
11 18 1 6 0 0 0
13 12 1 0 0 0 0
11 12 1 0 0 0 0
14 15 1 0 0 0 0
15 19 1 0 0 0 0
11 9 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 0 0 0 0
20 22 2 0 0 0 0
20 23 1 0 0 0 0
27 24 1 0 0 0 0
24 26 2 0 0 0 0
24 25 1 0 0 0 0
27 28 1 0 0 0 0
24 23 1 0 0 0 0
28 29 1 0 0 0 0
29 32 1 0 0 0 0
29 31 1 0 0 0 0
29 30 1 0 0 0 0
32 33 1 0 0 0 0
32 42 1 1 0 0 0
33 43 2 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
37 44 2 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
12 16 1 1 0 0 0
13 17 1 1 0 0 0
S SKP 7
ID BMCCPUAPf028
NAME Dephospho-CoA
FORMULA C21H35N7O13P2S
EXACTMASS 687.1488
AVERAGEMASS 687.5555
SMILES P(OP(OC[C@H]([C@H]3O)O[C@H]([C@@H]3O)n(c2)c(n1)c(n2)c(N)nc1)(O)=O)(OCC(C)(C)[C@H](C(=O)NCCC(=O)NCCS)O)(O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00882
M END