Mol:FL7AAIGL0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0727 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0727 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0727 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0727 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5164 0.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5164 0.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9601 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9601 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9601 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9601 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5164 1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5164 1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4038 0.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4038 0.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1525 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1525 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1525 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1525 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4038 1.5029 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.4038 1.5029 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7086 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7086 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2755 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2755 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8425 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8425 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8425 2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8425 2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2755 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2755 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7086 2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7086 2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6288 1.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6288 1.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7085 2.6575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7085 2.6575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5164 -0.4239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5164 -0.4239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5686 -0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5686 -0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0471 -1.4954 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.0471 -1.4954 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5400 -1.1078 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5400 -1.1078 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6712 -1.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6712 -1.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4965 -2.4038 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4965 -2.4038 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0471 -2.7218 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.0471 -2.7218 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8724 -2.1104 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8724 -2.1104 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.2218 -1.0906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2218 -1.0906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3739 -2.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3739 -2.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3707 -3.6680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3707 -3.6680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8915 -0.9931 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.8915 -0.9931 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.1950 -1.5851 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1950 -1.5851 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8037 -1.4640 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8037 -1.4640 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.3545 -1.5550 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.3545 -1.5550 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0255 -1.0850 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.0255 -1.0850 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.4807 -1.2602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4807 -1.2602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5730 -1.5063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5730 -1.5063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0335 -2.0695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0335 -2.0695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6338 -1.8877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6338 -1.8877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5927 -0.5744 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5927 -0.5744 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.2215 -1.0643 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2215 -1.0643 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6870 -0.8565 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6870 -0.8565 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1713 -0.8509 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1713 -0.8509 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5461 -0.4761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5461 -0.4761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0137 -0.7228 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.0137 -0.7228 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.1836 -0.5226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1836 -0.5226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5719 -1.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5719 -1.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3808 -1.3706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3808 -1.3706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7061 -0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7061 -0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2895 0.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2895 0.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5589 3.0610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5589 3.0610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0002 3.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0002 3.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2640 -0.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2640 -0.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0117 -0.4210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0117 -0.4210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8425 1.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8425 1.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8425 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8425 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1181 -2.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1181 -2.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3033 -3.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3033 -3.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 39 45 1 0 0 0 0 | + | 39 45 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 19 1 0 0 0 0 | + | 42 19 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 15 50 1 0 0 0 0 | + | 15 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 34 52 1 0 0 0 0 | + | 34 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 13 54 1 0 0 0 0 | + | 13 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 24 56 1 0 0 0 0 | + | 24 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 56 57 | + | M SAL 5 2 56 57 |
| − | M SBL 5 1 61 | + | M SBL 5 1 61 |
| − | M SMT 5 CH2OH | + | M SMT 5 CH2OH |
| − | M SVB 5 61 0.2057 -2.6485 | + | M SVB 5 61 0.2057 -2.6485 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 52 53 | + | M SAL 4 2 52 53 |
| − | M SBL 4 1 57 | + | M SBL 4 1 57 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 57 3.3097 -0.6593 | + | M SVB 4 57 3.3097 -0.6593 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 48 49 | + | M SAL 3 2 48 49 |
| − | M SBL 3 1 53 | + | M SBL 3 1 53 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 53 -3.4121 0.0613 | + | M SVB 3 53 -3.4121 0.0613 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 54 55 | + | M SAL 2 2 54 55 |
| − | M SBL 2 1 59 | + | M SBL 2 1 59 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 59 2.0521 1.2965 | + | M SVB 2 59 2.0521 1.2965 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 55 1.5589 3.061 | + | M SVB 1 55 1.5589 3.061 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGL0008 | + | ID FL7AAIGL0008 |
| − | KNApSAcK_ID C00006744 | + | KNApSAcK_ID C00006744 |
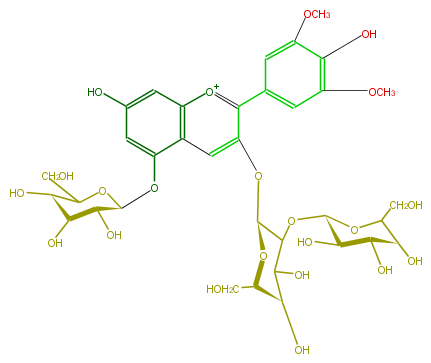
| − | NAME Malvidin 3-sophoroside-5-glucoside | + | NAME Malvidin 3-sophoroside-5-glucoside |
| − | CAS_RN 61811-04-9 | + | CAS_RN 61811-04-9 |
| − | FORMULA C35H45O22 | + | FORMULA C35H45O22 |
| − | EXACTMASS 817.240248124 | + | EXACTMASS 817.240248124 |
| − | AVERAGEMASS 817.7186 | + | AVERAGEMASS 817.7186 |
| − | SMILES O[C@H](C(CO)1)[C@@H](O)[C@@H](O)[C@@H](OC(C(O)2)[C@H](Oc(c6)c([o+1]c(c64)cc(O)cc(O[C@H](O5)[C@H]([C@@H](O)[C@H](C5CO)O)O)4)c(c3)cc(OC)c(c(OC)3)O)O[C@H](CO)[C@H](O)2)O1 | + | SMILES O[C@H](C(CO)1)[C@@H](O)[C@@H](O)[C@@H](OC(C(O)2)[C@H](Oc(c6)c([o+1]c(c64)cc(O)cc(O[C@H](O5)[C@H]([C@@H](O)[C@H](C5CO)O)O)4)c(c3)cc(OC)c(c(OC)3)O)O[C@H](CO)[C@H](O)2)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-2.0727 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0727 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5164 0.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9601 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9601 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5164 1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4038 0.2182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1525 0.5394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1525 1.1817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4038 1.5029 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.7086 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2755 1.1755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8425 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8425 2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2755 2.4848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7086 2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6288 1.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7085 2.6575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5164 -0.4239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5686 -0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0471 -1.4954 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5400 -1.1078 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6712 -1.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4965 -2.4038 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0471 -2.7218 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8724 -2.1104 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.2218 -1.0906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3739 -2.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3707 -3.6680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8915 -0.9931 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.1950 -1.5851 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8037 -1.4640 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.3545 -1.5550 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0255 -1.0850 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.4807 -1.2602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5730 -1.5063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0335 -2.0695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6338 -1.8877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5927 -0.5744 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.2215 -1.0643 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6870 -0.8565 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1713 -0.8509 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5461 -0.4761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0137 -0.7228 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.1836 -0.5226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5719 -1.5256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3808 -1.3706 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7061 -0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2895 0.4028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5589 3.0610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0002 3.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2640 -0.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0117 -0.4210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8425 1.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8425 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1181 -2.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3033 -3.3443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
27 30 1 0 0 0 0
22 20 1 0 0 0 0
39 40 1 1 0 0 0
40 41 1 1 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 39 1 0 0 0 0
39 45 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 19 1 0 0 0 0
44 48 1 0 0 0 0
48 49 1 0 0 0 0
15 50 1 0 0 0 0
50 51 1 0 0 0 0
34 52 1 0 0 0 0
52 53 1 0 0 0 0
13 54 1 0 0 0 0
54 55 1 0 0 0 0
24 56 1 0 0 0 0
56 57 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 56 57
M SBL 5 1 61
M SMT 5 CH2OH
M SVB 5 61 0.2057 -2.6485
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 52 53
M SBL 4 1 57
M SMT 4 CH2OH
M SVB 4 57 3.3097 -0.6593
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 48 49
M SBL 3 1 53
M SMT 3 CH2OH
M SVB 3 53 -3.4121 0.0613
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 54 55
M SBL 2 1 59
M SMT 2 OCH3
M SVB 2 59 2.0521 1.2965
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 55
M SMT 1 OCH3
M SVB 1 55 1.5589 3.061
S SKP 8
ID FL7AAIGL0008
KNApSAcK_ID C00006744
NAME Malvidin 3-sophoroside-5-glucoside
CAS_RN 61811-04-9
FORMULA C35H45O22
EXACTMASS 817.240248124
AVERAGEMASS 817.7186
SMILES O[C@H](C(CO)1)[C@@H](O)[C@@H](O)[C@@H](OC(C(O)2)[C@H](Oc(c6)c([o+1]c(c64)cc(O)cc(O[C@H](O5)[C@H]([C@@H](O)[C@H](C5CO)O)O)4)c(c3)cc(OC)c(c(OC)3)O)O[C@H](CO)[C@H](O)2)O1
M END