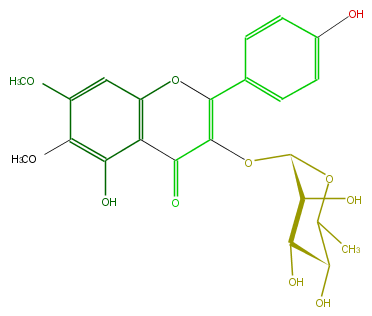
Mol:FL5FEAGS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 34 37 0 0 0 0 0 0 0 0999 V2000 | + | 34 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9987 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9987 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9987 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9987 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2976 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2976 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5965 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5965 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5965 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5965 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2976 1.5613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2976 1.5613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1046 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1046 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8057 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8057 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8057 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8057 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1046 1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1046 1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1046 -0.8958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1046 -0.8958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5066 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5066 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2212 1.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2212 1.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9356 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9356 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9356 2.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9356 2.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2212 2.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2212 2.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5066 2.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5066 2.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5729 -0.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5729 -0.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6501 2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6501 2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2976 -0.8672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2976 -0.8672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3969 -1.7156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3969 -1.7156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1886 -2.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1886 -2.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9373 -1.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9373 -1.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1886 -0.4092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1886 -0.4092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3969 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3969 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6479 -0.8312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6479 -0.8312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6189 -0.8312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6189 -0.8312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4572 -2.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4572 -2.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9975 -2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9975 -2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4936 -1.8082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4936 -1.8082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6143 -0.0085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6143 -0.0085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6501 -0.0994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6501 -0.0994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6563 1.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6563 1.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2865 2.6276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2865 2.6276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 2 31 1 0 0 0 0 | + | 2 31 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 1 33 1 0 0 0 0 | + | 1 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 35 0.6155 0.3554 | + | M SBV 1 35 0.6155 0.3554 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 33 34 | + | M SAL 2 2 33 34 |
| − | M SBL 2 1 37 | + | M SBL 2 1 37 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 37 0.6576 -0.3797 | + | M SBV 2 37 0.6576 -0.3797 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FEAGS0013 | + | ID FL5FEAGS0013 |
| − | FORMULA C23H24O11 | + | FORMULA C23H24O11 |
| − | EXACTMASS 476.13186161 | + | EXACTMASS 476.13186161 |
| − | AVERAGEMASS 476.43006 | + | AVERAGEMASS 476.43006 |
| − | SMILES OC(C1OC(=C(c(c4)ccc(c4)O)3)C(=O)c(c2O)c(O3)cc(c2OC)OC)C(C(O)C(C)O1)O | + | SMILES OC(C1OC(=C(c(c4)ccc(c4)O)3)C(=O)c(c2O)c(O3)cc(c2OC)OC)C(C(O)C(C)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
34 37 0 0 0 0 0 0 0 0999 V2000
-1.9987 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9987 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2976 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5965 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5965 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2976 1.5613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1046 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8057 0.3469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8057 1.1565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1046 1.5613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1046 -0.8958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5066 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2212 1.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9356 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9356 2.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2212 2.7988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5066 2.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5729 -0.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6501 2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2976 -0.8672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3969 -1.7156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1886 -2.1727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9373 -1.2936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1886 -0.4092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3969 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6479 -0.8312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6189 -0.8312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4572 -2.4759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9975 -2.8856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4936 -1.8082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6143 -0.0085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6501 -0.0994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6563 1.5361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2865 2.6276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
19 15 1 0 0 0 0
3 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
25 18 1 0 0 0 0
31 32 1 0 0 0 0
2 31 1 0 0 0 0
33 34 1 0 0 0 0
1 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 35
M SMT 1 ^ OCH3
M SBV 1 35 0.6155 0.3554
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 33 34
M SBL 2 1 37
M SMT 2 ^ OCH3
M SBV 2 37 0.6576 -0.3797
S SKP 5
ID FL5FEAGS0013
FORMULA C23H24O11
EXACTMASS 476.13186161
AVERAGEMASS 476.43006
SMILES OC(C1OC(=C(c(c4)ccc(c4)O)3)C(=O)c(c2O)c(O3)cc(c2OC)OC)C(C(O)C(C)O1)O
M END