Mol:FL5FDCGS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.4155 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4155 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7010 0.8310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7010 0.8310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7010 1.6560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7010 1.6560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4155 2.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4155 2.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1300 1.6561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1300 1.6561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1300 0.8311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1300 0.8311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9866 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9866 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2721 0.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2721 0.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5576 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5576 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5576 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5576 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2721 -0.8189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2721 -0.8189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9866 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9866 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8432 0.8310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8432 0.8310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1287 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1287 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1287 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1287 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8432 -0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8432 -0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2721 -1.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2721 -1.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5858 0.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5858 0.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7010 -0.8190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7010 -0.8190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8432 -1.6440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8432 -1.6440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8444 2.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8444 2.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4155 2.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4155 2.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4155 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4155 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7682 -0.9431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7682 -0.9431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9760 -1.1744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9760 -1.1744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5435 -0.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5435 -0.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8132 -0.0872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8132 -0.0872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6054 0.1443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6054 0.1443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0379 -0.5586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0379 -0.5586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7933 -0.4993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7933 -0.4993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4753 -1.0754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4753 -1.0754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8786 -1.6752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8786 -1.6752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3033 -1.5824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3033 -1.5824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6433 -0.5000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6433 -0.5000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0344 -2.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0344 -2.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8489 -2.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8489 -2.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0344 -1.2610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0344 -1.2610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4825 -1.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4825 -1.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3996 -1.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3996 -1.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8503 -2.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8503 -2.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4404 -1.6840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4404 -1.6840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3996 -2.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3996 -2.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8444 -2.8897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8444 -2.8897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 18 1 0 0 0 0 | + | 27 18 1 0 0 0 0 |
| − | 40 35 1 1 0 0 0 | + | 40 35 1 1 0 0 0 |
| − | 39 35 1 1 0 0 0 | + | 39 35 1 1 0 0 0 |
| − | 38 40 1 1 0 0 0 | + | 38 40 1 1 0 0 0 |
| − | 40 36 1 0 0 0 0 | + | 40 36 1 0 0 0 0 |
| − | 41 38 1 0 0 0 0 | + | 41 38 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FDCGS0013 | + | ID FL5FDCGS0013 |
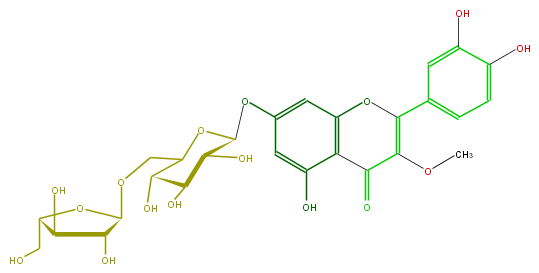
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES COC(C2=O)=C(Oc(c3)c(c(cc3OC(C(O)4)OC(COC(C(O)5)OC(CO)C5O)C(O)C4O)O)2)c(c1)ccc(O)c(O)1 | + | SMILES COC(C2=O)=C(Oc(c3)c(c(cc3OC(C(O)4)OC(COC(C(O)5)OC(CO)C5O)C(O)C4O)O)2)c(c1)ccc(O)c(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
4.4155 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7010 0.8310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7010 1.6560 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4155 2.0686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1300 1.6561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1300 0.8311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9866 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2721 0.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5576 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5576 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2721 -0.8189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9866 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8432 0.8310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1287 0.4185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1287 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8432 -0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2721 -1.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5858 0.8310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7010 -0.8190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8432 -1.6440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8444 2.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4155 2.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4155 -0.4065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7682 -0.9431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9760 -1.1744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5435 -0.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8132 -0.0872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6054 0.1443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0379 -0.5586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7933 -0.4993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4753 -1.0754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8786 -1.6752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3033 -1.5824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6433 -0.5000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0344 -2.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8489 -2.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0344 -1.2610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4825 -1.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3996 -1.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8503 -2.3027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4404 -1.6840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3996 -2.6329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8444 -2.8897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
19 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 18 1 0 0 0 0
40 35 1 1 0 0 0
39 35 1 1 0 0 0
38 40 1 1 0 0 0
40 36 1 0 0 0 0
41 38 1 0 0 0 0
39 41 1 0 0 0 0
35 37 1 0 0 0 0
39 42 1 0 0 0 0
42 43 1 0 0 0 0
31 38 1 0 0 0 0
S SKP 5
ID FL5FDCGS0013
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES COC(C2=O)=C(Oc(c3)c(c(cc3OC(C(O)4)OC(COC(C(O)5)OC(CO)C5O)C(O)C4O)O)2)c(c1)ccc(O)c(O)1
M END