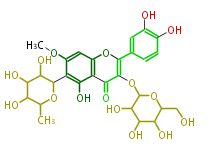
Mol:FL5FCCDS0001
From Metabolomics.JP
(Difference between revisions)
| (2 intermediate revisions by one user not shown) | |||
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3281 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3281 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6137 1.7362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6137 1.7362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1008 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1008 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1008 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1008 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6137 0.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6137 0.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3281 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3281 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8153 1.7362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8153 1.7362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5297 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5297 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5297 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5297 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8153 0.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8153 0.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1755 1.6965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1755 1.6965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8153 -0.6533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8153 -0.6533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6137 -0.4859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6137 -0.4859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1099 1.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1099 1.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8244 1.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8244 1.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5388 1.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5388 1.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5388 2.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5388 2.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8244 3.0125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8244 3.0125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1099 2.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1099 2.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1540 2.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1540 2.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9651 0.1309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9651 0.1309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2355 1.3728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2355 1.3728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8574 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8574 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2353 0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2353 0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0324 0.6767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0324 0.6767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3109 0.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3109 0.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8944 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8944 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0971 -0.2229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0971 -0.2229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8186 0.1779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8186 0.1779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3858 -0.8873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3858 -0.8873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1977 0.2618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1977 0.2618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2355 1.1867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2355 1.1867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8033 1.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8033 1.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4226 -2.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4226 -2.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2381 -2.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2381 -2.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3314 -1.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3314 -1.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8295 -0.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8295 -0.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0140 -0.8873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0140 -0.8873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9206 -1.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9206 -1.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3396 -1.9117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3396 -1.9117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1156 -2.4255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1156 -2.4255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2876 -3.0125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2876 -3.0125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8977 -2.7712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8977 -2.7712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0532 -1.1512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0532 -1.1512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 5 13 2 0 0 0 0 | + | 5 13 2 0 0 0 0 |
| − | 1 14 1 0 0 0 0 | + | 1 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 11 23 1 0 0 0 0 | + | 11 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 24 33 1 0 0 0 0 | + | 24 33 1 0 0 0 0 |
| − | 28 9 1 0 0 0 0 | + | 28 9 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 21 1 0 0 0 0 | + | 37 21 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID | + | ID FL5FCCDS0001 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES OC(C5CO)C(O)C(C(O5)OC(=C(c(c4)cc(c(O)c4)O)3)C(=O)c(c1O)c(O3)cc(OC)c1C(C2O)OC(C(C(O)2)O)C)O | + | SMILES OC(C5CO)C(O)C(C(O5)OC(=C(c(c4)cc(c(O)c4)O)3)C(=O)c(c1O)c(O3)cc(OC)c1C(C2O)OC(C(C(O)2)O)C)O |
M END | M END | ||
| − | |||
Latest revision as of 12:31, 30 September 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
1.3281 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6137 1.7362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1008 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1008 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6137 0.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3281 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8153 1.7362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5297 1.3237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5297 0.4987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8153 0.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1755 1.6965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8153 -0.6533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6137 -0.4859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1099 1.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8244 1.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5388 1.7750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5388 2.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8244 3.0125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1099 2.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1540 2.9552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9651 0.1309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2355 1.3728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8574 1.3028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2353 0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0324 0.6767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3109 0.2760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8944 -0.4364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0971 -0.2229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8186 0.1779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3858 -0.8873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1977 0.2618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2355 1.1867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8033 1.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4226 -2.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2381 -2.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3314 -1.4187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8295 -0.7607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0140 -0.8873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9206 -1.7074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3396 -1.9117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1156 -2.4255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2876 -3.0125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8977 -2.7712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0532 -1.1512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
8 11 1 0 0 0 0
10 12 1 0 0 0 0
5 13 2 0 0 0 0
1 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
17 20 1 0 0 0 0
6 21 1 0 0 0 0
16 22 1 0 0 0 0
11 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
27 30 1 0 0 0 0
26 31 1 0 0 0 0
25 32 1 0 0 0 0
24 33 1 0 0 0 0
28 9 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 21 1 0 0 0 0
S SKP 5
ID FL5FCCDS0001
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES OC(C5CO)C(O)C(C(O5)OC(=C(c(c4)cc(c(O)c4)O)3)C(=O)c(c1O)c(O3)cc(OC)c1C(C2O)OC(C(C(O)2)O)C)O
M END