Mol:FL5FCAGL0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5505 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5505 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5505 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5505 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9942 -0.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9942 -0.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4379 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4379 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4379 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4379 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9942 0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9942 0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8816 -0.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8816 -0.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3253 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3253 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3253 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3253 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8816 0.3836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8816 0.3836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8816 -1.4019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8816 -1.4019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7692 0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7692 0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2023 0.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2023 0.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3647 0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3647 0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3647 1.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3647 1.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2023 1.3655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2023 1.3655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7692 1.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7692 1.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8919 -1.0133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8919 -1.0133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9942 -1.5432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9942 -1.5432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9986 1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9986 1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9807 1.5982 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9807 1.5982 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.6799 1.0772 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.6799 1.0772 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2584 1.2425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2584 1.2425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8403 1.0772 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8403 1.0772 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1412 1.5982 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1412 1.5982 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5627 1.4329 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.5627 1.4329 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.8347 1.9996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8347 1.9996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6042 1.9075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6042 1.9075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5753 1.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5753 1.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4132 -0.9387 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.4132 -0.9387 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.1124 -1.4597 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.1124 -1.4597 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.6909 -1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6909 -1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2728 -1.4597 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.2728 -1.4597 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5737 -0.9387 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5737 -0.9387 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.9952 -1.1039 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9952 -1.1039 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.1455 -1.0884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1455 -1.0884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0368 -0.6294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0368 -0.6294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0078 -1.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0078 -1.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0399 0.7848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0399 0.7848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0399 -0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0399 -0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9078 0.6812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9078 0.6812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4078 1.5472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4078 1.5472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4724 -1.7521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4724 -1.7521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4724 -2.5771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4724 -2.5771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 31 18 1 0 0 0 0 | + | 31 18 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 39 1 0 0 0 0 | + | 24 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 33 43 1 0 0 0 0 | + | 33 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 43 44 | + | M SAL 3 2 43 44 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 47 1.6258 -1.433 | + | M SVB 3 47 1.6258 -1.433 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 39 40 | + | M SAL 2 2 39 40 |
| − | M SBL 2 1 43 | + | M SBL 2 1 43 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 43 3.1933 1.1039 | + | M SVB 2 43 3.1933 1.1039 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 45 -3.9078 0.6812 | + | M SVB 1 45 -3.9078 0.6812 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCAGL0005 | + | ID FL5FCAGL0005 |
| − | KNApSAcK_ID C00005281 | + | KNApSAcK_ID C00005281 |
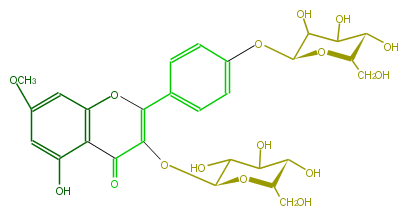
| − | NAME Rhamnocitrin 3,4'-diglucoside | + | NAME Rhamnocitrin 3,4'-diglucoside |
| − | CAS_RN 116183-66-5 | + | CAS_RN 116183-66-5 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES c(c5)(cc(O2)c(c(O)5)C(C(=C2c(c4)ccc(c4)O[C@H](O3)C(O)C(O)[C@@H](O)[C@H]3CO)O[C@H](O1)C(C(O)[C@H]([C@H]1CO)O)O)=O)OC | + | SMILES c(c5)(cc(O2)c(c(O)5)C(C(=C2c(c4)ccc(c4)O[C@H](O3)C(O)C(O)[C@@H](O)[C@H]3CO)O[C@H](O1)C(C(O)[C@H]([C@H]1CO)O)O)=O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.5505 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5505 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9942 -0.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4379 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4379 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9942 0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8816 -0.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3253 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3253 0.0624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8816 0.3836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8816 -1.4019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7692 0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2023 0.0562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3647 0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3647 1.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2023 1.3655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7692 1.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8919 -1.0133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9942 -1.5432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9986 1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9807 1.5982 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.6799 1.0772 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2584 1.2425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8403 1.0772 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1412 1.5982 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5627 1.4329 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.8347 1.9996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6042 1.9075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5753 1.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4132 -0.9387 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.1124 -1.4597 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.6909 -1.2944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2728 -1.4597 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5737 -0.9387 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.9952 -1.1039 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.1455 -1.0884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0368 -0.6294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0078 -1.1893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0399 0.7848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0399 -0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9078 0.6812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4078 1.5472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4724 -1.7521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4724 -2.5771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
31 18 1 0 0 0 0
22 20 1 0 0 0 0
24 39 1 0 0 0 0
39 40 1 0 0 0 0
1 41 1 0 0 0 0
41 42 1 0 0 0 0
33 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 43 44
M SBL 3 1 47
M SMT 3 CH2OH
M SVB 3 47 1.6258 -1.433
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 39 40
M SBL 2 1 43
M SMT 2 CH2OH
M SVB 2 43 3.1933 1.1039
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 OCH3
M SVB 1 45 -3.9078 0.6812
S SKP 8
ID FL5FCAGL0005
KNApSAcK_ID C00005281
NAME Rhamnocitrin 3,4'-diglucoside
CAS_RN 116183-66-5
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES c(c5)(cc(O2)c(c(O)5)C(C(=C2c(c4)ccc(c4)O[C@H](O3)C(O)C(O)[C@@H](O)[C@H]3CO)O[C@H](O1)C(C(O)[C@H]([C@H]1CO)O)O)=O)OC
M END