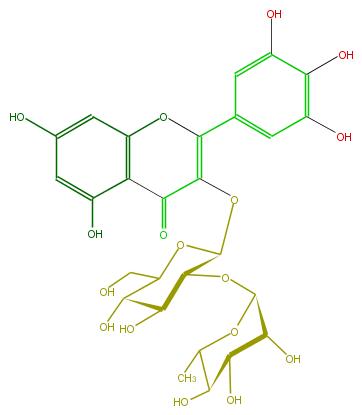
Mol:FL5FAGGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.7862 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7862 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 2.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 2.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7862 3.0596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7862 3.0596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 2.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 2.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5006 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5006 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 1.8221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 1.8221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0717 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0717 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0717 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0717 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3572 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3572 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7862 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7862 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5006 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5006 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5006 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5006 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7862 0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7862 0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3572 -0.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3572 -0.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2151 1.8221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2151 1.8221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0717 0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0717 0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7862 -0.5188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7862 -0.5188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2151 3.0596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2151 3.0596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7862 3.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7862 3.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1786 1.4308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1786 1.4308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1715 -1.8205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1715 -1.8205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3781 -2.0473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3781 -2.0473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0503 -1.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0503 -1.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7784 -0.9534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7784 -0.9534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0151 -0.7264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0151 -0.7264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4436 -1.4318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4436 -1.4318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0242 -1.3069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0242 -1.3069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5604 -1.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5604 -1.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5528 -2.3814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5528 -2.3814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0237 -2.4220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0237 -2.4220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8890 -1.3863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8890 -1.3863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3485 -2.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3485 -2.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 -3.6742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 -3.6742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9615 -2.9595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9615 -2.9595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6808 -2.5549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6808 -2.5549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4805 -1.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4805 -1.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0678 -2.4689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0678 -2.4689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1677 -3.0166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1677 -3.0166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9873 -3.8429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9873 -3.8429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0652 -3.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0652 -3.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0033 -3.3978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0033 -3.3978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 35 44 1 0 0 0 0 | + | 35 44 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0023 | + | ID FL5FAGGS0023 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES C(C5O)(OC(C)C(C5O)O)OC(C1OC(=C(c(c4)cc(c(c(O)4)O)O)3)C(c(c2O3)c(cc(c2)O)O)=O)C(O)C(C(CO)O1)O | + | SMILES C(C5O)(OC(C)C(C5O)O)OC(C1OC(=C(c(c4)cc(c(c(O)4)O)O)3)C(c(c2O3)c(cc(c2)O)O)=O)C(O)C(C(CO)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
1.7862 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 2.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7862 3.0596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 2.6471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5006 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 1.8221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0717 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0717 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7862 1.8221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5006 1.4096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5006 0.5846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7862 0.1721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 -0.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2151 1.8221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0717 0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7862 -0.5188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2151 3.0596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7862 3.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1786 1.4308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1715 -1.8205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3781 -2.0473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0503 -1.3420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7784 -0.9534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0151 -0.7264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4436 -1.4318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0242 -1.3069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5604 -1.6696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5528 -2.3814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0237 -2.4220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8890 -1.3863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3485 -2.8736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 -3.6742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9615 -2.9595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6808 -2.5549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4805 -1.7542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0678 -2.4689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1677 -3.0166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9873 -3.8429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0652 -3.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0033 -3.3978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
37 42 1 0 0 0 0
36 43 1 0 0 0 0
35 44 1 0 0 0 0
39 34 1 0 0 0 0
27 19 1 0 0 0 0
S SKP 5
ID FL5FAGGS0023
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES C(C5O)(OC(C)C(C5O)O)OC(C1OC(=C(c(c4)cc(c(c(O)4)O)O)3)C(c(c2O3)c(cc(c2)O)O)=O)C(O)C(C(CO)O1)O
M END