Mol:FL5FADNSS005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 37 0 0 0 0 0 0 0 0999 V2000 | + | 35 37 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8150 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8150 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8150 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8150 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2587 -1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2587 -1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7024 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7024 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7024 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7024 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2587 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2587 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1461 -1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1461 -1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4102 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4102 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4102 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4102 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1461 0.1250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1461 0.1250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1461 -1.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1461 -1.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9663 0.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9663 0.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5333 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5333 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1003 0.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1003 0.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1003 0.7796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1003 0.7796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5333 1.1069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5333 1.1069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9663 0.7796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9663 0.7796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2581 -1.7018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2581 -1.7018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4544 0.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4544 0.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7567 -1.3545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7567 -1.3545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6172 -2.2149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6172 -2.2149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1692 -1.7586 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1692 -1.7586 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5702 -1.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5702 -1.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7848 -2.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7848 -2.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9663 1.2796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9663 1.2796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7483 1.5634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7483 1.5634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1664 1.2986 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1664 1.2986 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9595 1.7423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9595 1.7423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3962 0.8058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3962 0.8058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7950 0.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7950 0.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1143 0.2223 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1143 0.2223 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1143 0.7118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1143 0.7118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1143 -0.3215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1143 -0.3215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8263 1.6886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8263 1.6886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2762 2.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2762 2.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 3 1 0 0 0 0 | + | 18 3 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 22 24 2 0 0 0 0 | + | 22 24 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 27 29 2 0 0 0 0 | + | 27 29 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 15 25 1 0 0 0 0 | + | 15 25 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 2 0 0 0 0 | + | 31 33 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 19 31 1 0 0 0 0 | + | 19 31 1 0 0 0 0 |
| − | 16 34 1 0 0 0 0 | + | 16 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 34 35 | + | M SAL 1 2 34 35 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 36 1.8263 1.6886 | + | M SVB 1 36 1.8263 1.6886 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADNSS005 | + | ID FL5FADNSS005 |
| − | KNApSAcK_ID C00004972 | + | KNApSAcK_ID C00004972 |
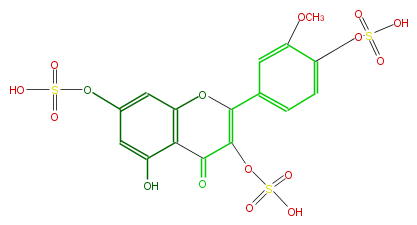
| − | NAME Isorhamnetin 3,7,4'-tri-O-sulfate | + | NAME Isorhamnetin 3,7,4'-tri-O-sulfate |
| − | CAS_RN 79175-00-1 | + | CAS_RN 79175-00-1 |
| − | FORMULA C16H12O16S3 | + | FORMULA C16H12O16S3 |
| − | EXACTMASS 555.928746406 | + | EXACTMASS 555.928746406 |
| − | AVERAGEMASS 556.45488 | + | AVERAGEMASS 556.45488 |
| − | SMILES COc(c1)c(ccc1C(O2)=C(OS(O)(=O)=O)C(=O)c(c(O)3)c(cc(c3)OS(O)(=O)=O)2)OS(O)(=O)=O | + | SMILES COc(c1)c(ccc1C(O2)=C(OS(O)(=O)=O)C(=O)c(c(O)3)c(cc(c3)OS(O)(=O)=O)2)OS(O)(=O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 37 0 0 0 0 0 0 0 0999 V2000
-1.8150 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8150 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2587 -1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7024 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7024 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2587 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1461 -1.1597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4102 -0.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4102 -0.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1461 0.1250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1461 -1.6605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9663 0.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5333 -0.2024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1003 0.1249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1003 0.7796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5333 1.1069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9663 0.7796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2581 -1.7018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4544 0.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7567 -1.3545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6172 -2.2149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1692 -1.7586 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
1.5702 -1.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7848 -2.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9663 1.2796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7483 1.5634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1664 1.2986 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
2.9595 1.7423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3962 0.8058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7950 0.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1143 0.2223 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
-3.1143 0.7118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1143 -0.3215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8263 1.6886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2762 2.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 3 1 0 0 0 0
1 19 1 0 0 0 0
22 23 2 0 0 0 0
22 24 2 0 0 0 0
21 22 1 0 0 0 0
20 22 1 0 0 0 0
8 20 1 0 0 0 0
27 28 2 0 0 0 0
27 29 2 0 0 0 0
26 27 1 0 0 0 0
25 27 1 0 0 0 0
15 25 1 0 0 0 0
31 32 2 0 0 0 0
31 33 2 0 0 0 0
30 31 1 0 0 0 0
19 31 1 0 0 0 0
16 34 1 0 0 0 0
34 35 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 34 35
M SBL 1 1 36
M SMT 1 OCH3
M SVB 1 36 1.8263 1.6886
S SKP 8
ID FL5FADNSS005
KNApSAcK_ID C00004972
NAME Isorhamnetin 3,7,4'-tri-O-sulfate
CAS_RN 79175-00-1
FORMULA C16H12O16S3
EXACTMASS 555.928746406
AVERAGEMASS 556.45488
SMILES COc(c1)c(ccc1C(O2)=C(OS(O)(=O)=O)C(=O)c(c(O)3)c(cc(c3)OS(O)(=O)=O)2)OS(O)(=O)=O
M END