Mol:FL5FACGL0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5360 -2.5535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5360 -2.5535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0440 -2.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0440 -2.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4403 -2.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4403 -2.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3288 -2.1141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3288 -2.1141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8208 -1.7012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8208 -1.7012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4245 -1.9209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4245 -1.9209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7252 -1.8944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7252 -1.8944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6136 -1.2618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6136 -1.2618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1057 -0.8489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1057 -0.8489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7093 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7093 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3414 -2.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3414 -2.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9942 -0.2165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9942 -0.2165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3789 0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3789 0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2653 0.6522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2653 0.6522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7668 1.0730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7668 1.0730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3820 0.8490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3820 0.8490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4957 0.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4957 0.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1394 -2.7731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1394 -2.7731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9485 -3.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9485 -3.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6907 1.5047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6907 1.5047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2518 -0.3195 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.2518 -0.3195 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.9386 -0.8620 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9386 -0.8620 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5409 -0.6899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5409 -0.6899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1469 -0.8620 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1469 -0.8620 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4602 -0.3195 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4602 -0.3195 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.8578 -0.4916 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.8578 -0.4916 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.7099 -0.3195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7099 -0.3195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9010 0.0026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9010 0.0026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1467 -0.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1467 -0.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6674 -0.6193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6674 -0.6193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7660 0.8828 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.7660 0.8828 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.1720 1.2663 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.1720 1.2663 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6760 0.8314 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.6760 0.8314 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1210 0.6227 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1210 0.6227 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6734 0.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6734 0.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1864 0.7914 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.1864 0.7914 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 5.2376 1.0091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2376 1.0091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0818 1.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0818 1.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1430 1.2668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1430 1.2668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9830 -0.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9830 -0.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2443 -1.1669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2443 -1.1669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8833 1.2697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8833 1.2697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3936 0.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3936 0.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0716 -0.5934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0716 -0.5934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 47 1.3688 -1.1907 | + | M SVB 1 47 1.3688 -1.1907 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0011 | + | ID FL5FACGL0011 |
| − | KNApSAcK_ID C00005411 | + | KNApSAcK_ID C00005411 |
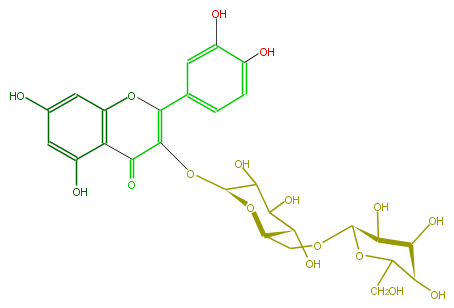
| − | NAME Quercetin 3-gentiobioside | + | NAME Quercetin 3-gentiobioside |
| − | CAS_RN 7431-83-6 | + | CAS_RN 7431-83-6 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)O[C@H](O1)C(C(O)[C@H]([C@@H](CO[C@@H]([C@H]2O)OC([C@@H]([C@H](O)2)O)CO)1)O)O | + | SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)O[C@H](O1)C(C(O)[C@H]([C@@H](CO[C@@H]([C@H]2O)OC([C@@H]([C@H](O)2)O)CO)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.5360 -2.5535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0440 -2.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4403 -2.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3288 -2.1141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8208 -1.7012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4245 -1.9209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7252 -1.8944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6136 -1.2618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1057 -0.8489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7093 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3414 -2.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9942 -0.2165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3789 0.0074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2653 0.6522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7668 1.0730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3820 0.8490 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4957 0.2043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1394 -2.7731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9485 -3.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6907 1.5047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2518 -0.3195 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.9386 -0.8620 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5409 -0.6899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1469 -0.8620 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4602 -0.3195 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.8578 -0.4916 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.7099 -0.3195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9010 0.0026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1467 -0.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6674 -0.6193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7660 0.8828 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.1720 1.2663 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6760 0.8314 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1210 0.6227 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6734 0.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1864 0.7914 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
5.2376 1.0091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0818 1.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1430 1.2668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9830 -0.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2443 -1.1669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8833 1.2697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3936 0.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0716 -0.5934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
16 42 1 0 0 0 0
36 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 47
M SMT 1 CH2OH
M SVB 1 47 1.3688 -1.1907
S SKP 8
ID FL5FACGL0011
KNApSAcK_ID C00005411
NAME Quercetin 3-gentiobioside
CAS_RN 7431-83-6
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)O[C@H](O1)C(C(O)[C@H]([C@@H](CO[C@@H]([C@H]2O)OC([C@@H]([C@H](O)2)O)CO)1)O)O
M END