Mol:FL5FAAGS0138
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 77 84 0 0 0 0 0 0 0 0999 V2000 | + | 77 84 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.1197 4.4141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1197 4.4141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1197 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1197 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8342 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8342 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5487 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5487 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5487 4.4141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5487 4.4141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8342 4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8342 4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4052 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4052 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6908 3.5891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6908 3.5891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9763 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9763 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9763 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9763 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6908 1.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6908 1.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4052 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4052 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2618 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2618 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5474 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5474 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5474 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5474 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2618 1.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2618 1.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6908 1.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6908 1.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1671 3.5891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1671 3.5891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0829 1.9168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0829 1.9168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1494 4.7609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1494 4.7609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2618 1.2395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2618 1.2395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8668 3.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8668 3.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3605 2.7108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3605 2.7108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5982 3.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5982 3.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7811 2.9106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7811 2.9106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2874 3.5623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2874 3.5623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0498 3.2462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0498 3.2462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2703 3.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2703 3.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6960 4.0920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6960 4.0920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2445 2.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2445 2.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7557 2.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7557 2.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1610 2.4643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1610 2.4643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7717 2.8013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7717 2.8013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2654 2.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2654 2.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5031 2.4658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5031 2.4658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6860 2.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6860 2.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1923 3.0012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1923 3.0012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9547 2.6850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9547 2.6850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1752 3.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1752 3.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6009 3.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6009 3.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1494 2.3029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1494 2.3029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6607 2.1992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6607 2.1992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0659 1.9031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0659 1.9031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7897 -0.3599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7897 -0.3599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6150 -0.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6150 -0.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8315 0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8315 0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4236 1.0129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4236 1.0129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5983 1.0114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5983 1.0114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3817 0.2150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3817 0.2150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7956 0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7956 0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3163 0.0929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3163 0.0929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9679 -1.0196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9679 -1.0196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1668 -0.6182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1668 -0.6182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3009 -0.0295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3009 -0.0295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0641 -2.4876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0641 -2.4876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8877 -2.4356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8877 -2.4356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0552 -1.6275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0552 -1.6275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6111 -1.0175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6111 -1.0175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7874 -1.0694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7874 -1.0694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6198 -1.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6198 -1.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0253 -1.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0253 -1.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5638 -2.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5638 -2.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2536 -3.1949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2536 -3.1949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4998 -2.6595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4998 -2.6595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5522 -2.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5522 -2.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4005 -3.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4005 -3.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8568 -4.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8568 -4.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5766 -3.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5766 -3.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1646 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1646 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3408 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3408 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9283 -3.3976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9283 -3.3976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1033 -3.3976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1033 -3.3976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6908 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6908 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1033 -4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1033 -4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9283 -4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9283 -4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1331 -4.1121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1331 -4.1121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6913 -2.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6913 -2.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 30 1 0 0 0 0 | + | 36 30 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 52 1 0 0 0 0 | + | 44 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 19 1 0 0 0 0 | + | 47 19 1 0 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 58 57 1 1 0 0 0 | + | 58 57 1 1 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 55 63 1 0 0 0 0 | + | 55 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 57 65 1 0 0 0 0 | + | 57 65 1 0 0 0 0 |
| − | 58 54 1 0 0 0 0 | + | 58 54 1 0 0 0 0 |
| − | 66 67 2 0 0 0 0 | + | 66 67 2 0 0 0 0 |
| − | 66 68 1 0 0 0 0 | + | 66 68 1 0 0 0 0 |
| − | 68 69 2 0 0 0 0 | + | 68 69 2 0 0 0 0 |
| − | 69 70 1 0 0 0 0 | + | 69 70 1 0 0 0 0 |
| − | 70 71 2 0 0 0 0 | + | 70 71 2 0 0 0 0 |
| − | 71 72 1 0 0 0 0 | + | 71 72 1 0 0 0 0 |
| − | 72 73 2 0 0 0 0 | + | 72 73 2 0 0 0 0 |
| − | 73 74 1 0 0 0 0 | + | 73 74 1 0 0 0 0 |
| − | 74 75 2 0 0 0 0 | + | 74 75 2 0 0 0 0 |
| − | 75 70 1 0 0 0 0 | + | 75 70 1 0 0 0 0 |
| − | 73 76 1 0 0 0 0 | + | 73 76 1 0 0 0 0 |
| − | 72 77 1 0 0 0 0 | + | 72 77 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGS0138 | + | ID FL5FAAGS0138 |
| − | KNApSAcK_ID C00013805 | + | KNApSAcK_ID C00013805 |
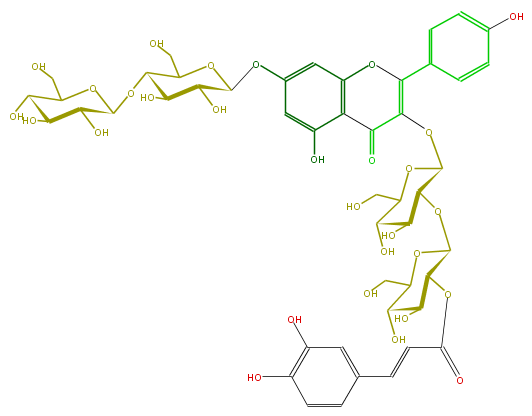
| − | NAME Kaempferol 3-(2'''-(E)-caffeoylglucosyl)-(1->2)-glucoside-7-cellobioside;3-[[2-O-[2-O-[(2E)-3-(3,4-Dihydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]-beta-D-glucopyranosyl]oxy]-7-[(4-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Kaempferol 3-(2'''-(E)-caffeoylglucosyl)-(1->2)-glucoside-7-cellobioside;3-[[2-O-[2-O-[(2E)-3-(3,4-Dihydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]-beta-D-glucopyranosyl]oxy]-7-[(4-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 220342-45-0 | + | CAS_RN 220342-45-0 |
| − | FORMULA C48H56O29 | + | FORMULA C48H56O29 |
| − | EXACTMASS 1096.29072583 | + | EXACTMASS 1096.29072583 |
| − | AVERAGEMASS 1096.94084 | + | AVERAGEMASS 1096.94084 |
| − | SMILES C(O)(C(O)2)C(C(OC2Oc(c8)cc(O)c(c83)C(C(OC(C(OC(C6OC(=O)C=Cc(c7)ccc(c7O)O)OC(C(C6O)O)CO)5)OC(CO)C(C(O)5)O)=C(c(c4)ccc(c4)O)O3)=O)CO)OC(C1O)OC(C(O)C(O)1)CO | + | SMILES C(O)(C(O)2)C(C(OC2Oc(c8)cc(O)c(c83)C(C(OC(C(OC(C6OC(=O)C=Cc(c7)ccc(c7O)O)OC(C(C6O)O)CO)5)OC(CO)C(C(O)5)O)=C(c(c4)ccc(c4)O)O3)=O)CO)OC(C1O)OC(C(O)C(O)1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
77 84 0 0 0 0 0 0 0 0999 V2000
4.1197 4.4141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1197 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8342 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5487 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5487 4.4141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8342 4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4052 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6908 3.5891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9763 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9763 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6908 1.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4052 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2618 3.5891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5474 3.1766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5474 2.3516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2618 1.9391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6908 1.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1671 3.5891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0829 1.9168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1494 4.7609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2618 1.2395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8668 3.3625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3605 2.7108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5982 3.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7811 2.9106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2874 3.5623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0498 3.2462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2703 3.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6960 4.0920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2445 2.8641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7557 2.7604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1610 2.4643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7717 2.8013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2654 2.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5031 2.4658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6860 2.3494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1923 3.0012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9547 2.6850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1752 3.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6009 3.5309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1494 2.3029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6607 2.1992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0659 1.9031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7897 -0.3599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6150 -0.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8315 0.4380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4236 1.0129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5983 1.0114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3817 0.2150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7956 0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3163 0.0929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9679 -1.0196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1668 -0.6182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3009 -0.0295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0641 -2.4876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8877 -2.4356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0552 -1.6275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6111 -1.0175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7874 -1.0694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6198 -1.8775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0253 -1.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5638 -2.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2536 -3.1949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4998 -2.6595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5522 -2.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4005 -3.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8568 -4.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5766 -3.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1646 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3408 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9283 -3.3976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1033 -3.3976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6908 -4.1121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1033 -4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9283 -4.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1331 -4.1121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6913 -2.6841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 30 1 0 0 0 0
18 25 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
44 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 19 1 0 0 0 0
55 56 1 1 0 0 0
56 57 1 1 0 0 0
58 57 1 1 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
60 55 1 0 0 0 0
60 61 1 0 0 0 0
61 62 1 0 0 0 0
55 63 1 0 0 0 0
56 64 1 0 0 0 0
57 65 1 0 0 0 0
58 54 1 0 0 0 0
66 67 2 0 0 0 0
66 68 1 0 0 0 0
68 69 2 0 0 0 0
69 70 1 0 0 0 0
70 71 2 0 0 0 0
71 72 1 0 0 0 0
72 73 2 0 0 0 0
73 74 1 0 0 0 0
74 75 2 0 0 0 0
75 70 1 0 0 0 0
73 76 1 0 0 0 0
72 77 1 0 0 0 0
65 66 1 0 0 0 0
S SKP 8
ID FL5FAAGS0138
KNApSAcK_ID C00013805
NAME Kaempferol 3-(2'''-(E)-caffeoylglucosyl)-(1->2)-glucoside-7-cellobioside;3-[[2-O-[2-O-[(2E)-3-(3,4-Dihydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]-beta-D-glucopyranosyl]oxy]-7-[(4-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 220342-45-0
FORMULA C48H56O29
EXACTMASS 1096.29072583
AVERAGEMASS 1096.94084
SMILES C(O)(C(O)2)C(C(OC2Oc(c8)cc(O)c(c83)C(C(OC(C(OC(C6OC(=O)C=Cc(c7)ccc(c7O)O)OC(C(C6O)O)CO)5)OC(CO)C(C(O)5)O)=C(c(c4)ccc(c4)O)O3)=O)CO)OC(C1O)OC(C(O)C(O)1)CO
M END