Mol:FL5FAAGS0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.8028 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8028 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8028 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8028 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4433 0.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4433 0.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0839 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0839 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0839 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0839 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4433 1.6047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4433 1.6047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7244 0.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7244 0.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3650 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3650 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3650 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3650 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7244 1.6047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7244 1.6047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7244 -0.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7244 -0.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0053 1.6045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0053 1.6045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6582 1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6582 1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3111 1.6045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3111 1.6045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3111 2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3111 2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6582 2.7353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6582 2.7353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0053 2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0053 2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4433 -0.6141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4433 -0.6141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1625 1.6045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1625 1.6045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9637 2.7352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9637 2.7352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1351 -0.0910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1351 -0.0910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2947 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2947 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3887 -0.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3887 -0.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8145 0.5532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8145 0.5532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1045 0.9733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1045 0.9733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9234 1.1929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9234 1.1929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3739 0.4771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3739 0.4771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6248 -0.4992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6248 -0.4992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9807 -0.4798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9807 -0.4798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0030 0.5988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0030 0.5988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8908 -2.0153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8908 -2.0153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9848 -2.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9848 -2.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4106 -1.6817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4106 -1.6817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7007 -1.2615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7007 -1.2615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5196 -1.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5196 -1.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9699 -1.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9699 -1.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1784 -2.6117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1784 -2.6117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5356 -2.7353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5356 -2.7353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6150 -1.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6150 -1.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8131 -1.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8131 -1.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9637 -1.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9637 -1.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 28 1 0 0 0 0 | + | 34 28 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 45 0.8432 -0.1796 | + | M SBV 1 45 0.8432 -0.1796 |
| − | S SKP 5 | + | S SKP 5 |
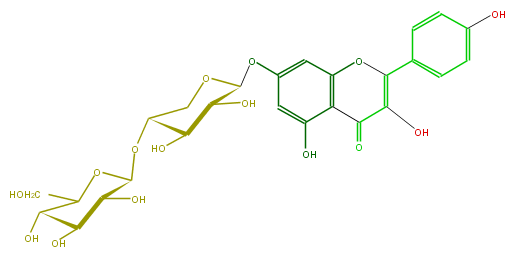
| − | ID FL5FAAGS0034 | + | ID FL5FAAGS0034 |
| − | FORMULA C26H28O15 | + | FORMULA C26H28O15 |
| − | EXACTMASS 580.1428202259999 | + | EXACTMASS 580.1428202259999 |
| − | AVERAGEMASS 580.49152 | + | AVERAGEMASS 580.49152 |
| − | SMILES Oc(c5)ccc(c5)C(=C(O)1)Oc(c2)c(c(O)cc2OC(C(O)4)OCC(C4O)OC(O3)C(C(C(C3CO)O)O)O)C1=O | + | SMILES Oc(c5)ccc(c5)C(=C(O)1)Oc(c2)c(c(O)cc2OC(C(O)4)OCC(C4O)OC(O3)C(C(C(C3CO)O)O)O)C1=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
0.8028 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8028 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4433 0.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0839 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0839 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4433 1.6047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7244 0.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3650 0.4951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3650 1.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7244 1.6047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7244 -0.4514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0053 1.6045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6582 1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3111 1.6045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3111 2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6582 2.7353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0053 2.3583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4433 -0.6141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1625 1.6045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9637 2.7352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1351 -0.0910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2947 0.2196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3887 -0.1622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8145 0.5532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1045 0.9733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9234 1.1929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3739 0.4771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6248 -0.4992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9807 -0.4798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0030 0.5988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8908 -2.0153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9848 -2.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4106 -1.6817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7007 -1.2615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5196 -1.0419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9699 -1.7578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1784 -2.6117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5356 -2.7353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6150 -1.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8131 -1.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9637 -1.5348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 28 1 0 0 0 0
25 19 1 0 0 0 0
40 41 1 0 0 0 0
36 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^ CH2OH
M SBV 1 45 0.8432 -0.1796
S SKP 5
ID FL5FAAGS0034
FORMULA C26H28O15
EXACTMASS 580.1428202259999
AVERAGEMASS 580.49152
SMILES Oc(c5)ccc(c5)C(=C(O)1)Oc(c2)c(c(O)cc2OC(C(O)4)OCC(C4O)OC(O3)C(C(C(C3CO)O)O)O)C1=O
M END