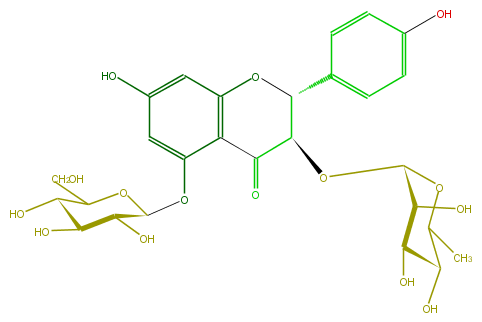
Mol:FL4DAAGS0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4705 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4705 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7561 0.0491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7561 0.0491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0416 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0416 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0416 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0416 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7561 1.6990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7561 1.6990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4705 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4705 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6728 0.0491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6728 0.0491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3872 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3872 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3872 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3872 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6728 1.6990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6728 1.6990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6728 -0.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6728 -0.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1823 1.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1823 1.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9075 1.2785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9075 1.2785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6326 1.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6326 1.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6326 2.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6326 2.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9075 2.9532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9075 2.9532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1823 2.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1823 2.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2017 1.7088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2017 1.7088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0294 -0.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0294 -0.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3577 2.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3577 2.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7561 -0.7751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7561 -0.7751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3111 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3111 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8392 -1.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8392 -1.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1596 -1.1101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1596 -1.1101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5039 -1.1030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5039 -1.1030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9803 -0.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9803 -0.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6746 -0.8756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6746 -0.8756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0404 -1.0503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0404 -1.0503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5309 -1.4054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5309 -1.4054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5818 -1.5397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5818 -1.5397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6204 -1.7301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6204 -1.7301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3547 -2.1541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3547 -2.1541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1217 -1.3388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1217 -1.3388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3547 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3547 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6204 -0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6204 -0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8533 -0.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8533 -0.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7727 -0.9565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7727 -0.9565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6176 -2.4138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6176 -2.4138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0831 -2.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0831 -2.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7037 -1.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7037 -1.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4454 -0.3513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4454 -0.3513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7727 -0.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7727 -0.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 35 19 1 0 0 0 0 | + | 35 19 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.7708 -0.5243 | + | M SBV 1 46 0.7708 -0.5243 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL4DAAGS0010 | + | ID FL4DAAGS0010 |
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES O=C(C1OC(C(O)5)OC(C)C(C5O)O)c(c3OC(O4)C(C(O)C(C4CO)O)O)c(cc(O)c3)OC1c(c2)ccc(c2)O | + | SMILES O=C(C1OC(C(O)5)OC(C)C(C5O)O)c(c3OC(O4)C(C(O)C(C4CO)O)O)c(cc(O)c3)OC1c(c2)ccc(c2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.4705 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7561 0.0491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0416 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0416 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7561 1.6990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4705 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6728 0.0491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3872 0.4616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3872 1.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6728 1.6990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6728 -0.6695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1823 1.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9075 1.2785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6326 1.6972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6326 2.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9075 2.9532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1823 2.5346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2017 1.7088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0294 -0.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3577 2.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7561 -0.7751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3111 -0.7513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8392 -1.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1596 -1.1101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5039 -1.1030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9803 -0.6264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6746 -0.8756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0404 -1.0503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5309 -1.4054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5818 -1.5397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6204 -1.7301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3547 -2.1541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1217 -1.3388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3547 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6204 -0.0944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8533 -0.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7727 -0.9565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6176 -2.4138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0831 -2.9532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7037 -1.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4454 -0.3513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7727 -0.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 21 1 0 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 31 1 1 0 0 0
36 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
35 19 1 0 0 0 0
41 42 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.7708 -0.5243
S SKP 5
ID FL4DAAGS0010
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES O=C(C1OC(C(O)5)OC(C)C(C5O)O)c(c3OC(O4)C(C(O)C(C4CO)O)O)c(cc(O)c3)OC1c(c2)ccc(c2)O
M END