Mol:FL3FAIDS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1103 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1103 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1103 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1103 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4056 -2.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4056 -2.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2991 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2991 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2991 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2991 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4056 -0.4447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4056 -0.4447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0037 -2.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0037 -2.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7084 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7084 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7084 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7084 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0037 -0.4447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0037 -0.4447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0037 -2.8078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0037 -2.8078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4056 -2.8855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4056 -2.8855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5697 -0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5697 -0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3123 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3123 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0548 -0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0548 -0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0548 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0548 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3123 0.9719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3123 0.9719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5697 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5697 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8173 0.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8173 0.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9192 -0.4060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9192 -0.4060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0329 -2.1677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0329 -2.1677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2127 -2.7052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2127 -2.7052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6831 -2.1576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6831 -2.1576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7981 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7981 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5525 -1.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5525 -1.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1460 -2.1464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1460 -2.1464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5355 -2.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5355 -2.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9764 -2.8287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9764 -2.8287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9057 -2.5040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9057 -2.5040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9608 -0.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9608 -0.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3076 -1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3076 -1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6545 -0.6151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6545 -0.6151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7661 -0.7196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7661 -0.7196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3932 -0.1447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3932 -0.1447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0986 -0.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0986 -0.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6260 -0.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6260 -0.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1405 -0.8860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1405 -0.8860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9312 -1.1714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9312 -1.1714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8252 -1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8252 -1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6437 -2.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6437 -2.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3123 1.7541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3123 1.7541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8819 2.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8819 2.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6950 0.1558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6950 0.1558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8960 0.5586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8960 0.5586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7989 -0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7989 -0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8960 -1.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8960 -1.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 17 41 1 0 0 0 0 | + | 17 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 15 45 1 0 0 0 0 | + | 15 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 44 0.6791 -0.4283 | + | M SBV 1 44 0.6791 -0.4283 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 46 0.0000 -0.7822 | + | M SBV 2 46 0.0000 -0.7822 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 43 44 | + | M SAL 3 2 43 44 |
| − | M SBL 3 1 48 | + | M SBL 3 1 48 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 48 0.5964 -0.6402 | + | M SBV 3 48 0.5964 -0.6402 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 45 46 | + | M SAL 4 2 45 46 |
| − | M SBL 4 1 50 | + | M SBL 4 1 50 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SBV 4 50 -0.7441 0.4296 | + | M SBV 4 50 -0.7441 0.4296 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAIDS0001 | + | ID FL3FAIDS0001 |
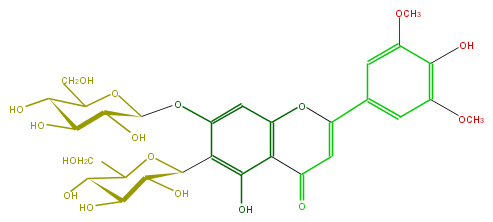
| − | FORMULA C29H34O17 | + | FORMULA C29H34O17 |
| − | EXACTMASS 654.179599662 | + | EXACTMASS 654.179599662 |
| − | AVERAGEMASS 654.57006 | + | AVERAGEMASS 654.57006 |
| − | SMILES C(C1O)(c(c(OC(C(O)5)OC(CO)C(O)C(O)5)4)c(O)c(c(c4)2)C(=O)C=C(c(c3)cc(OC)c(c3OC)O)O2)OC(CO)C(O)C1O | + | SMILES C(C1O)(c(c(OC(C(O)5)OC(CO)C(O)C(O)5)4)c(O)c(c(c4)2)C(=O)C=C(c(c3)cc(OC)c(c3OC)O)O2)OC(CO)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-1.1103 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1103 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4056 -2.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2991 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2991 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4056 -0.4447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0037 -2.0720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7084 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7084 -0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0037 -0.4447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0037 -2.8078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4056 -2.8855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5697 -0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3123 -0.7430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0548 -0.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0548 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3123 0.9719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5697 0.5433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8173 0.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9192 -0.4060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0329 -2.1677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2127 -2.7052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6831 -2.1576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7981 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5525 -1.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1460 -2.1464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5355 -2.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9764 -2.8287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9057 -2.5040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9608 -0.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3076 -1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6545 -0.6151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7661 -0.7196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3932 -0.1447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0986 -0.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6260 -0.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1405 -0.8860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9312 -1.1714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8252 -1.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6437 -2.7473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3123 1.7541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8819 2.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6950 0.1558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8960 0.5586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7989 -0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8960 -1.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 2 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 20 1 0 0 0 0
39 40 1 0 0 0 0
26 39 1 0 0 0 0
41 42 1 0 0 0 0
17 41 1 0 0 0 0
43 44 1 0 0 0 0
35 43 1 0 0 0 0
45 46 1 0 0 0 0
15 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ CH2OH
M SBV 1 44 0.6791 -0.4283
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 OCH3
M SBV 2 46 0.0000 -0.7822
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 43 44
M SBL 3 1 48
M SMT 3 ^ CH2OH
M SBV 3 48 0.5964 -0.6402
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 45 46
M SBL 4 1 50
M SMT 4 OCH3
M SBV 4 50 -0.7441 0.4296
S SKP 5
ID FL3FAIDS0001
FORMULA C29H34O17
EXACTMASS 654.179599662
AVERAGEMASS 654.57006
SMILES C(C1O)(c(c(OC(C(O)5)OC(CO)C(O)C(O)5)4)c(O)c(c(c4)2)C(=O)C=C(c(c3)cc(OC)c(c3OC)O)O2)OC(CO)C(O)C1O
M END