Mol:FL2FAAGS0027
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4707 0.2868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4707 0.2868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2419 0.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2419 0.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4676 -0.5382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4676 -0.5382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2492 -0.9475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2492 -0.9475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9618 -0.5318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9618 -0.5318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9582 0.2932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9582 0.2932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6781 -0.9412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6781 -0.9412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3908 -0.5255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3908 -0.5255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3871 0.2995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3871 0.2995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6708 0.7088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6708 0.7088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0409 0.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0409 0.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7570 0.2706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7570 0.2706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4698 0.6858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4698 0.6858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4666 1.5108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4666 1.5108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7506 1.9206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7506 1.9206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0377 1.5053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0377 1.5053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6792 -1.5844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6792 -1.5844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0962 0.6479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0962 0.6479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2492 -1.6721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2492 -1.6721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0854 1.8681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0854 1.8681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8369 1.1816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8369 1.1816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4242 0.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4242 0.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6259 0.6761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6259 0.6761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8324 0.4492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8324 0.4492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2450 1.1640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2450 1.1640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0434 0.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0434 0.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2767 0.0712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2767 0.0712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9599 0.5281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9599 0.5281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5090 0.9368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5090 0.9368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2597 1.3343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2597 1.3343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9611 1.4903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9611 1.4903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0854 1.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0854 1.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7982 -0.6080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7982 -0.6080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2108 -1.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2108 -1.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4173 -1.0959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4173 -1.0959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6190 -1.3051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6190 -1.3051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2063 -0.5905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2063 -0.5905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9999 -0.8172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9999 -0.8172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6237 -1.9206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6237 -1.9206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8715 -1.6991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8715 -1.6991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6659 -1.1312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6659 -1.1312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4089 -0.6290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4089 -0.6290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 27 1 0 0 0 0 | + | 37 27 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
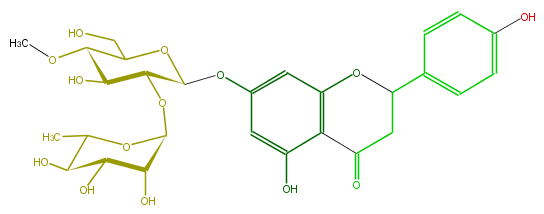
| − | ID FL2FAAGS0027 | + | ID FL2FAAGS0027 |
| − | FORMULA C28H34O14 | + | FORMULA C28H34O14 |
| − | EXACTMASS 594.194855796 | + | EXACTMASS 594.194855796 |
| − | AVERAGEMASS 594.56116 | + | AVERAGEMASS 594.56116 |
| − | SMILES c(c3OC(O5)C(C(O)C(OC)C5CO)OC(O4)C(O)C(C(C4C)O)O)c(O1)c(c(O)c3)C(=O)CC1c(c2)ccc(O)c2 | + | SMILES c(c3OC(O5)C(C(O)C(OC)C5CO)OC(O4)C(O)C(C(C4C)O)O)c(O1)c(c(O)c3)C(=O)CC1c(c2)ccc(O)c2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.4707 0.2868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2419 0.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4676 -0.5382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2492 -0.9475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9618 -0.5318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9582 0.2932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6781 -0.9412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3908 -0.5255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3871 0.2995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6708 0.7088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0409 0.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7570 0.2706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4698 0.6858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4666 1.5108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7506 1.9206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0377 1.5053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6792 -1.5844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0962 0.6479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2492 -1.6721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0854 1.8681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8369 1.1816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4242 0.4669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6259 0.6761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8324 0.4492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2450 1.1640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0434 0.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2767 0.0712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9599 0.5281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5090 0.9368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2597 1.3343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9611 1.4903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0854 1.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7982 -0.6080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2108 -1.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4173 -1.0959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6190 -1.3051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2063 -0.5905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9999 -0.8172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6237 -1.9206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8715 -1.6991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6659 -1.1312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4089 -0.6290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
14 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
23 27 1 0 0 0 0
22 28 1 0 0 0 0
21 29 1 0 0 0 0
26 30 1 0 0 0 0
24 18 1 0 0 0 0
31 30 1 0 0 0 0
29 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 27 1 0 0 0 0
S SKP 5
ID FL2FAAGS0027
FORMULA C28H34O14
EXACTMASS 594.194855796
AVERAGEMASS 594.56116
SMILES c(c3OC(O5)C(C(O)C(OC)C5CO)OC(O4)C(O)C(C(C4C)O)O)c(O1)c(c(O)c3)C(=O)CC1c(c2)ccc(O)c2
M END